Arvores de Regressão
Bibliotecas
Carregando os dados
#> [1] "crim" "zn" "indus" "chas" "nox" "rm" "age"
#> [8] "dis" "rad" "tax" "ptratio" "black" "lstat" "medv"describe(Boston)#> vars n mean sd median trimmed mad min max range skew
#> crim 1 506 3.61 8.60 0.26 1.68 0.33 0.01 88.98 88.97 5.19
#> zn 2 506 11.36 23.32 0.00 5.08 0.00 0.00 100.00 100.00 2.21
#> indus 3 506 11.14 6.86 9.69 10.93 9.37 0.46 27.74 27.28 0.29
#> chas 4 506 0.07 0.25 0.00 0.00 0.00 0.00 1.00 1.00 3.39
#> nox 5 506 0.55 0.12 0.54 0.55 0.13 0.38 0.87 0.49 0.72
#> rm 6 506 6.28 0.70 6.21 6.25 0.51 3.56 8.78 5.22 0.40
#> age 7 506 68.57 28.15 77.50 71.20 28.98 2.90 100.00 97.10 -0.60
#> dis 8 506 3.80 2.11 3.21 3.54 1.91 1.13 12.13 11.00 1.01
#> rad 9 506 9.55 8.71 5.00 8.73 2.97 1.00 24.00 23.00 1.00
#> tax 10 506 408.24 168.54 330.00 400.04 108.23 187.00 711.00 524.00 0.67
#> ptratio 11 506 18.46 2.16 19.05 18.66 1.70 12.60 22.00 9.40 -0.80
#> black 12 506 356.67 91.29 391.44 383.17 8.09 0.32 396.90 396.58 -2.87
#> lstat 13 506 12.65 7.14 11.36 11.90 7.11 1.73 37.97 36.24 0.90
#> medv 14 506 22.53 9.20 21.20 21.56 5.93 5.00 50.00 45.00 1.10
#> kurtosis se
#> crim 36.60 0.38
#> zn 3.95 1.04
#> indus -1.24 0.30
#> chas 9.48 0.01
#> nox -0.09 0.01
#> rm 1.84 0.03
#> age -0.98 1.25
#> dis 0.46 0.09
#> rad -0.88 0.39
#> tax -1.15 7.49
#> ptratio -0.30 0.10
#> black 7.10 4.06
#> lstat 0.46 0.32
#> medv 1.45 0.41Conhecendo as variáveis
crim: taxa de criminalidade per capita por cidade.
zn: proporção de terrenos residenciais zoneados para lotes acima de 25.000 pés quadrados.
indus: proporção de acres de negócios não varejistas por cidade.
chas: variável fictícia do rio Charles (= 1 se o trato limita o rio; 0 caso contrário). nox concentração de óxidos de nitrogênio (partes por 10 milhões).
rm: número médio de cômodos por habitação.
age: proporção de unidades ocupadas pelo proprietário construídas antes de 1940.
dis: média ponderada das distâncias até cinco centros de emprego de Boston.
rad: índice de acessibilidade a rodovias radiais. tax taxa de imposto sobre a propriedade do valor total por $ 10.000.
ptratio: proporção aluno-professor por cidade.
black: 1000(Bk−0,63)21000(Bk−0,63)2 onde BkBk é a proporção de negros por cidade.
lstat: status inferior da população (por cento).
medv: valor médio das casas ocupadas pelo proprietário em $ 1000s.
Selecionando os dados
#> medv nox rm
#> Min. : 5.00 Min. :0.3850 Min. :3.561
#> 1st Qu.:17.02 1st Qu.:0.4490 1st Qu.:5.886
#> Median :21.20 Median :0.5380 Median :6.208
#> Mean :22.53 Mean :0.5547 Mean :6.285
#> 3rd Qu.:25.00 3rd Qu.:0.6240 3rd Qu.:6.623
#> Max. :50.00 Max. :0.8710 Max. :8.780boxplot(extrato$medv)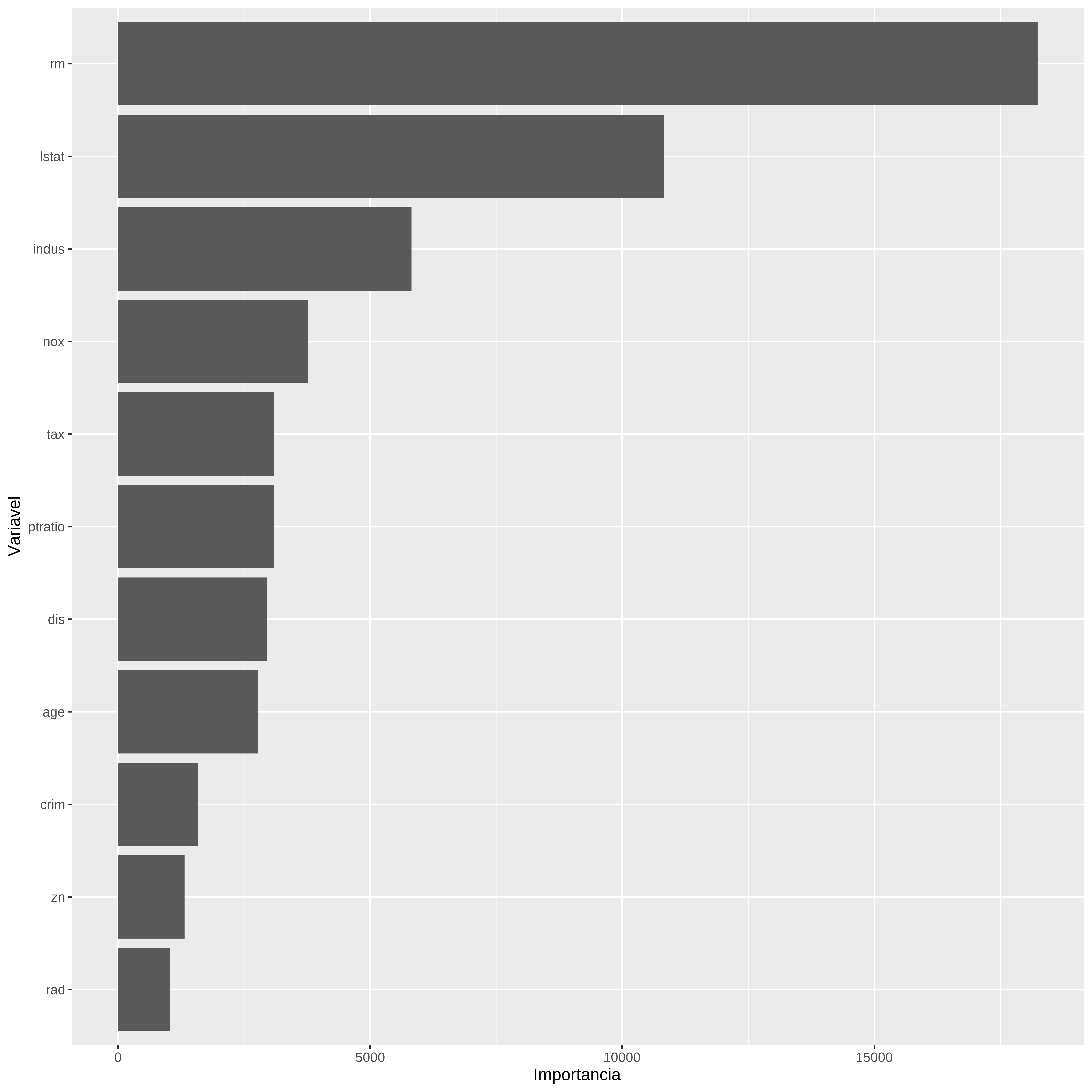
Visualizando os dados
## Distribuição de dados na maior parte simétrica com valores na cauda direta
## maior do que o esperado para uam distribuição simétrica
ggplot(extrato, aes(x=medv)) +
geom_histogram(bins = round(1+3.322*log10(nrow(extrato)),0))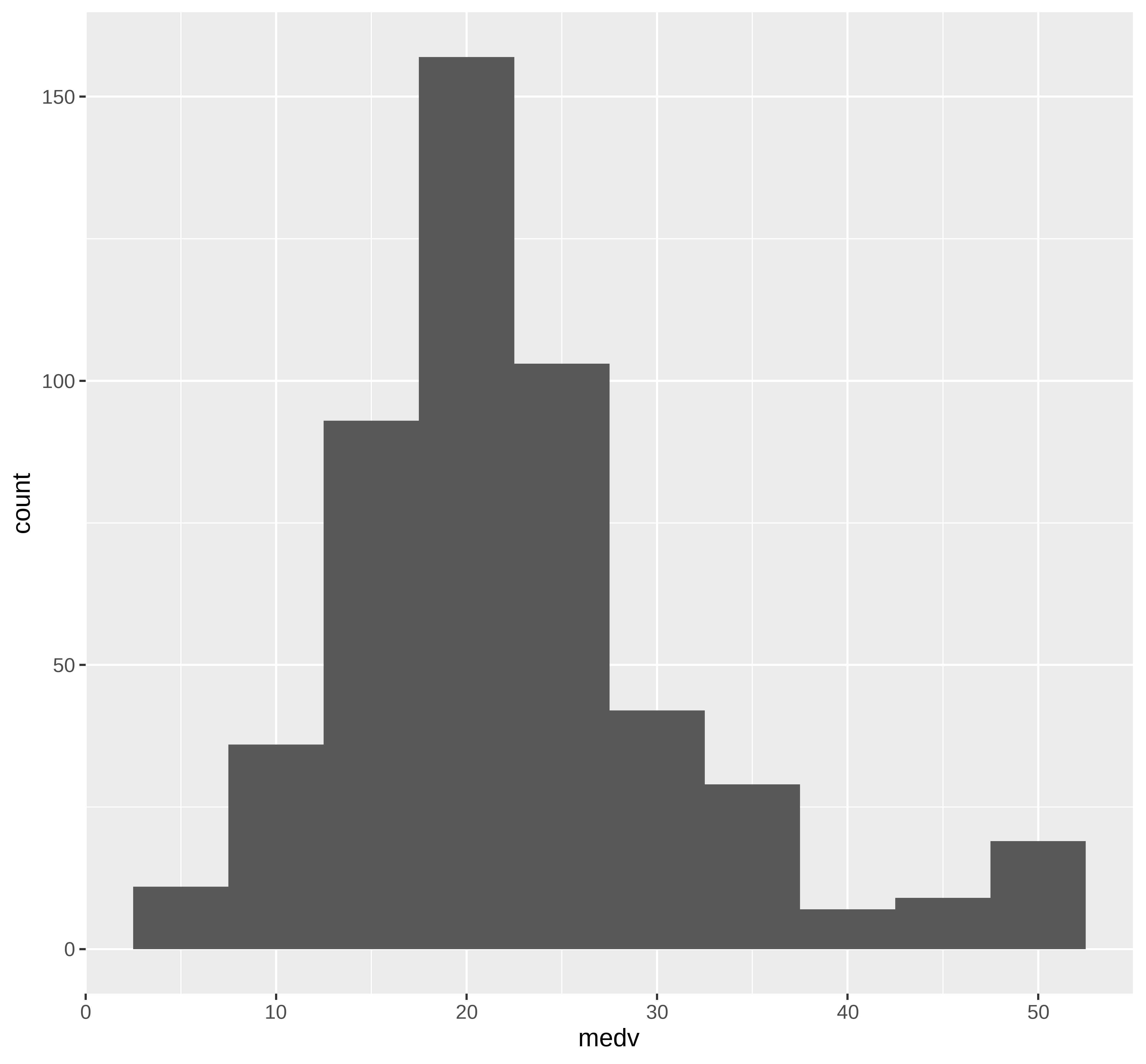
## Grafico de dispersão nox vs rm
ggplot(extrato, aes(x=rm, y=nox)) +
geom_point()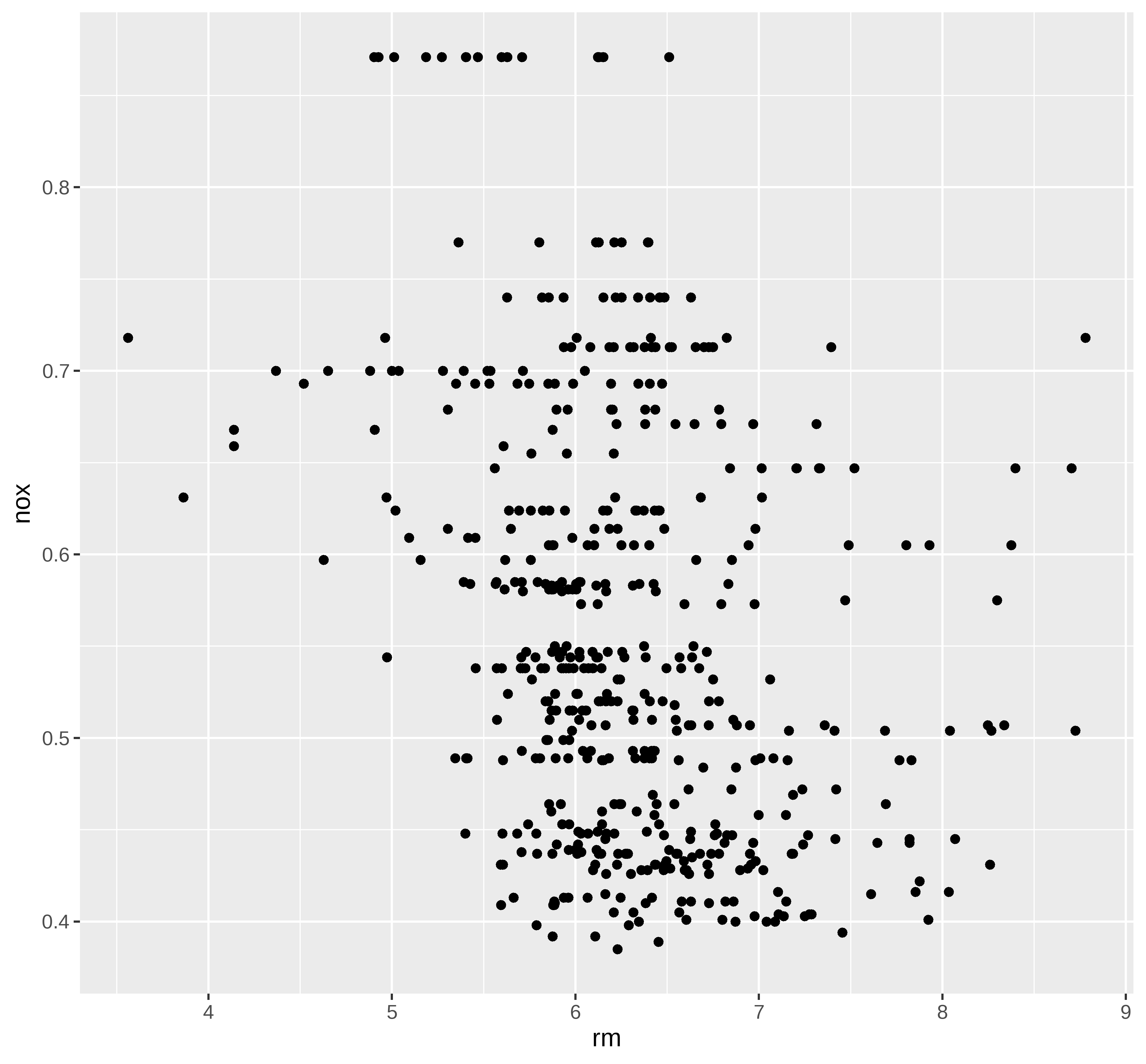
Arvore de Regressão
Na biblioteca rpart as arvores de regressão são obtidas usando o método anova. Existem alguns controles que podem ser feitos nos parametros da arvore.
Neste exemplo só definimos o menor conjunto de dados numa partição (minsplit) e o parametro de complexidade cp. Qualquer partição/divisão que não melhore o ajuste por um fator de cp não é tentada. Por exemplo, com a partição pela anova, isso significa que o R-quadrado geral deve aumentar pelo valor de cp a cada etapa. O principal papel deste parâmetro é economizar tempo de computação podando divisões que obviamente não valem a pena. Essencialmente, o usuário informa ao programa que qualquer divisão que não melhore o ajuste pelo cp, provavelmente será podada por validação cruzada, e que, portanto, não é necessário persegui-lo.
##Usando rpart para desenvolver a arvore
arvreg <- rpart(medv ~ .,
data=extrato,
method="anova", #para arvore de regressão
control=rpart.control(minsplit=30,cp=0.06))
arvreg#> n= 506
#>
#> node), split, n, deviance, yval
#> * denotes terminal node
#>
#> 1) root 506 42716.300 22.53281
#> 2) rm< 6.941 430 17317.320 19.93372
#> 4) nox>=0.6695 97 2214.391 13.73918 *
#> 5) nox< 0.6695 333 10296.590 21.73814 *
#> 3) rm>=6.941 76 6059.419 37.23816
#> 6) rm< 7.437 46 1899.612 32.11304 *
#> 7) rm>=7.437 30 1098.850 45.09667 *rpart.plot(arvreg)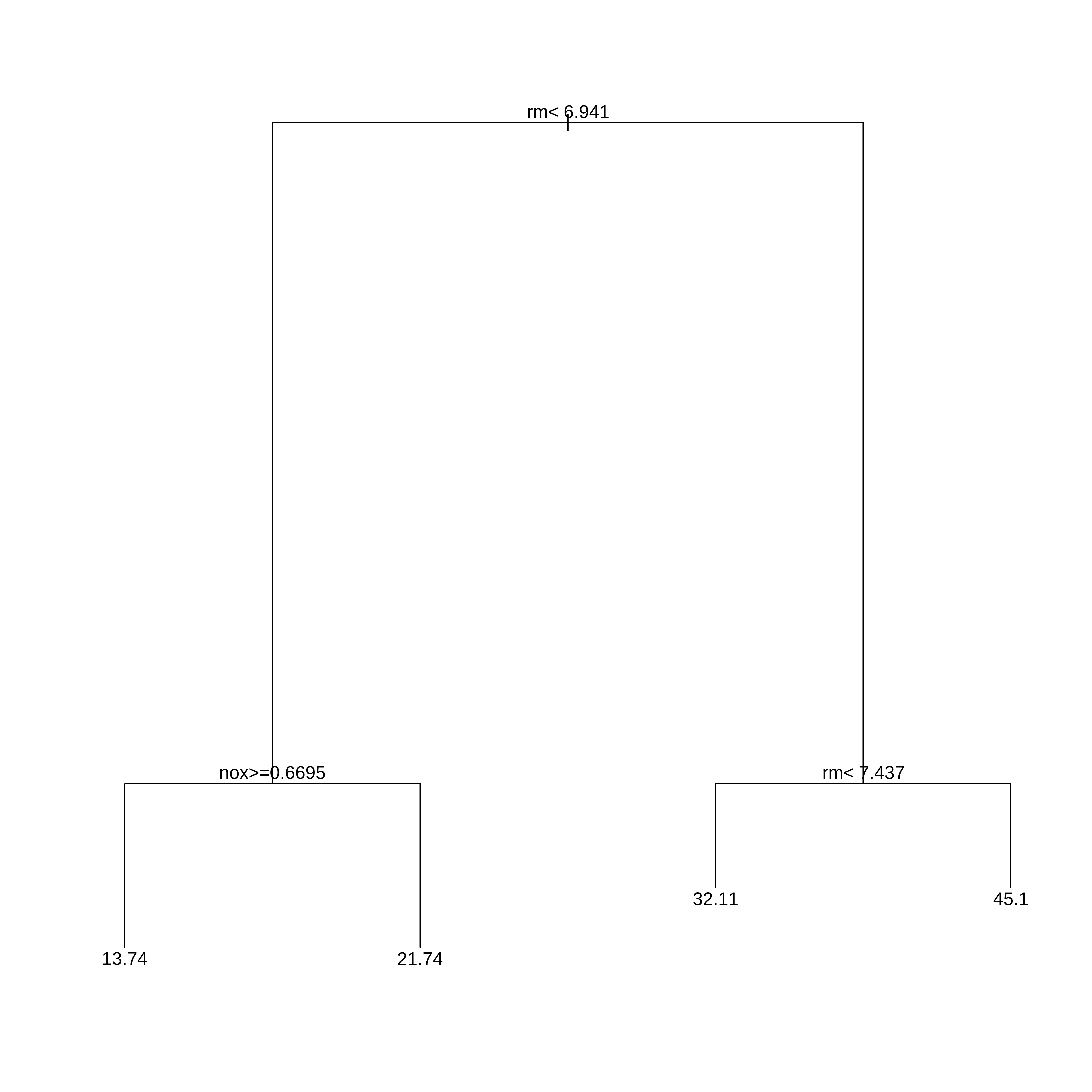
Segmentos
A partir da árvore obtida no item anterior podemos fazer uma representação gráfica das partições obtidas.
ggplot(extrato, aes(x=rm, y=nox)) +
geom_point() +
geom_segment(aes(x = 0, y = 0.6695, xend = 6.941, yend = 0.6695),
linetype="dashed", color="red", size=1) +
geom_vline(xintercept = 6.941, linetype="dashed", color="red", size=1) +
geom_vline(xintercept = 7.437, linetype="dashed", color="red", size=1) 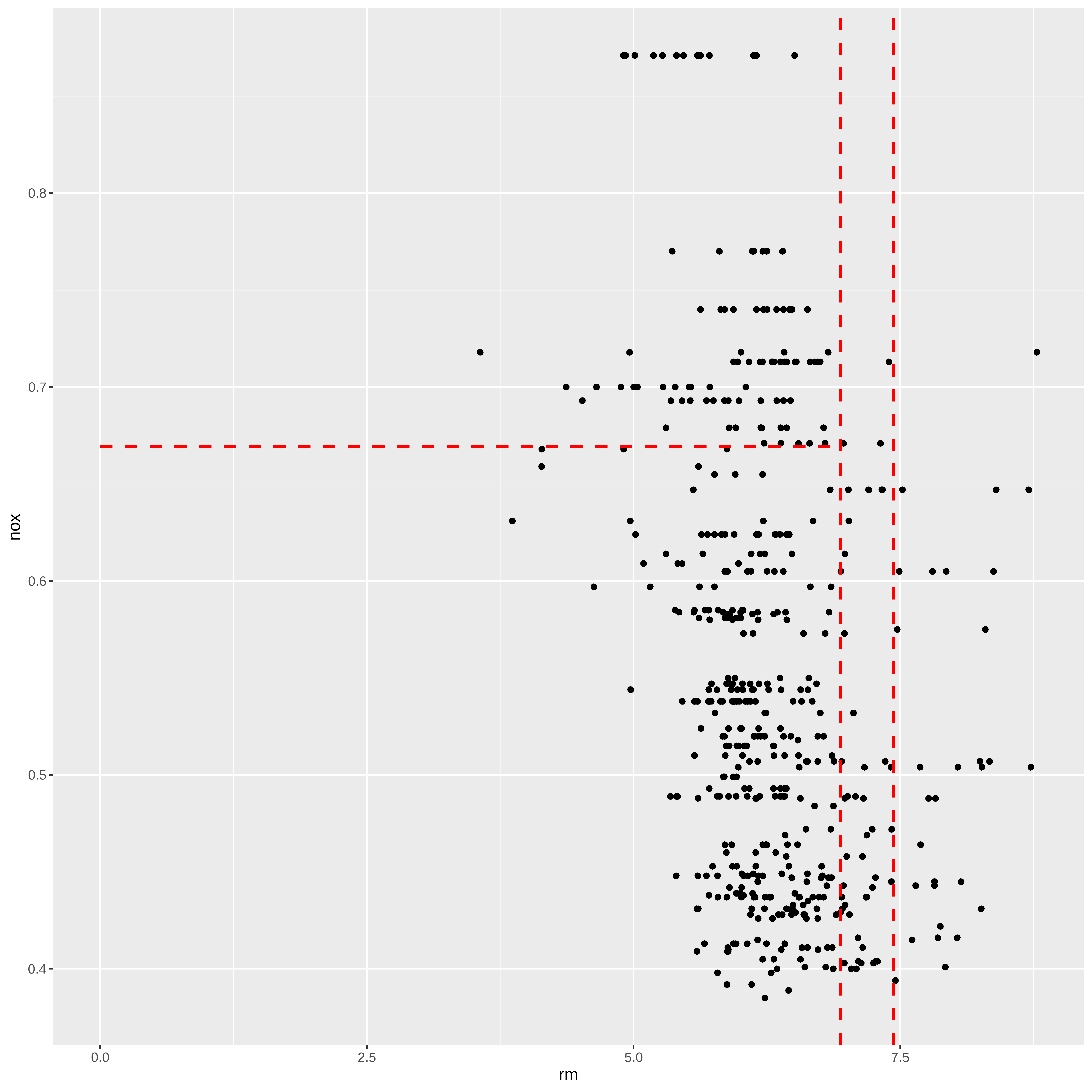
# scale_y_continuous(limits = c(0.3, 1)) +Treino e Teste com todas as variáveis
Agora vamos trabalhar com o conjunto completo criando um conjunto de treino e teste.
## Vamos criar os conjuntos de treino teste e desenvolver a arvore
## com todas as variáveis.
set.seed(2025)
indice <- createDataPartition(dados$medv, times=1, p=0.75, list=FALSE)
conj_treino <- dados[indice,]
conj_teste <- dados[-indice,]
head(conj_treino)#> crim zn indus chas nox rm age dis rad tax ptratio black lstat
#> 2 0.02731 0.0 7.07 0 0.469 6.421 78.9 4.9671 2 242 17.8 396.90 9.14
#> 6 0.02985 0.0 2.18 0 0.458 6.430 58.7 6.0622 3 222 18.7 394.12 5.21
#> 7 0.08829 12.5 7.87 0 0.524 6.012 66.6 5.5605 5 311 15.2 395.60 12.43
#> 8 0.14455 12.5 7.87 0 0.524 6.172 96.1 5.9505 5 311 15.2 396.90 19.15
#> 10 0.17004 12.5 7.87 0 0.524 6.004 85.9 6.5921 5 311 15.2 386.71 17.10
#> 11 0.22489 12.5 7.87 0 0.524 6.377 94.3 6.3467 5 311 15.2 392.52 20.45
#> medv
#> 2 21.6
#> 6 28.7
#> 7 22.9
#> 8 27.1
#> 10 18.9
#> 11 15.0head(conj_teste)#> crim zn indus chas nox rm age dis rad tax ptratio black
#> 1 0.00632 18.0 2.31 0 0.538 6.575 65.2 4.0900 1 296 15.3 396.90
#> 3 0.02729 0.0 7.07 0 0.469 7.185 61.1 4.9671 2 242 17.8 392.83
#> 4 0.03237 0.0 2.18 0 0.458 6.998 45.8 6.0622 3 222 18.7 394.63
#> 5 0.06905 0.0 2.18 0 0.458 7.147 54.2 6.0622 3 222 18.7 396.90
#> 9 0.21124 12.5 7.87 0 0.524 5.631 100.0 6.0821 5 311 15.2 386.63
#> 19 0.80271 0.0 8.14 0 0.538 5.456 36.6 3.7965 4 307 21.0 288.99
#> lstat medv
#> 1 4.98 24.0
#> 3 4.03 34.7
#> 4 2.94 33.4
#> 5 5.33 36.2
#> 9 29.93 16.5
#> 19 11.69 20.2Arvore de Regressão com caret
Aqui vamos usar a biblioteca caret que tem umas facilidades para otimização do cp e apresentação dos resultados
set.seed(2025)
## Otimizamos o valor de cp usando um 10-fold cv
# O parametro tuneLength diz para o algoritmo escolher diferentes valores para cp
# O parametro tuneGrid permite decidir que valores cp deve assumir enquanto que o
# tuneLength somente limita o número default de parametros que se usa.
arvreg2 <- train(medv ~ . , data = conj_treino, method = "rpart",
trControl = trainControl("cv", number = 10),
tuneGrid = data.frame(cp = seq(0.01,0.10, length.out=100))
)
# Mostra a acurácia vs cp (parametro de complexidade)
plot(arvreg2)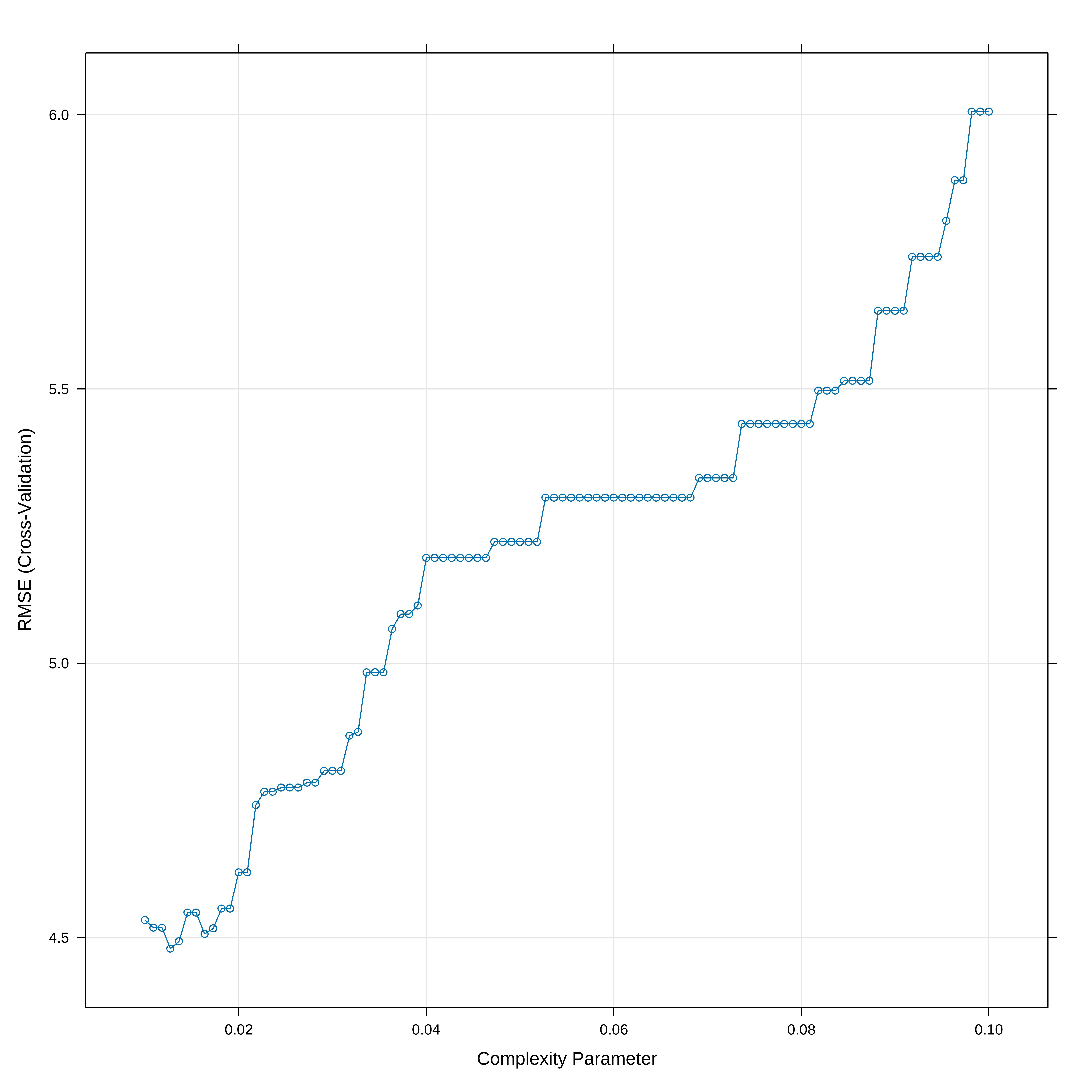
## Indica o melhor valor de cp
arvreg2$bestTune#> cp
#> 2 0.01090909Desenhando a Árvore
## Apresenta o modelo final de arvore ajustado
## usando o rpart.plot
rpart.plot(arvreg2$finalModel)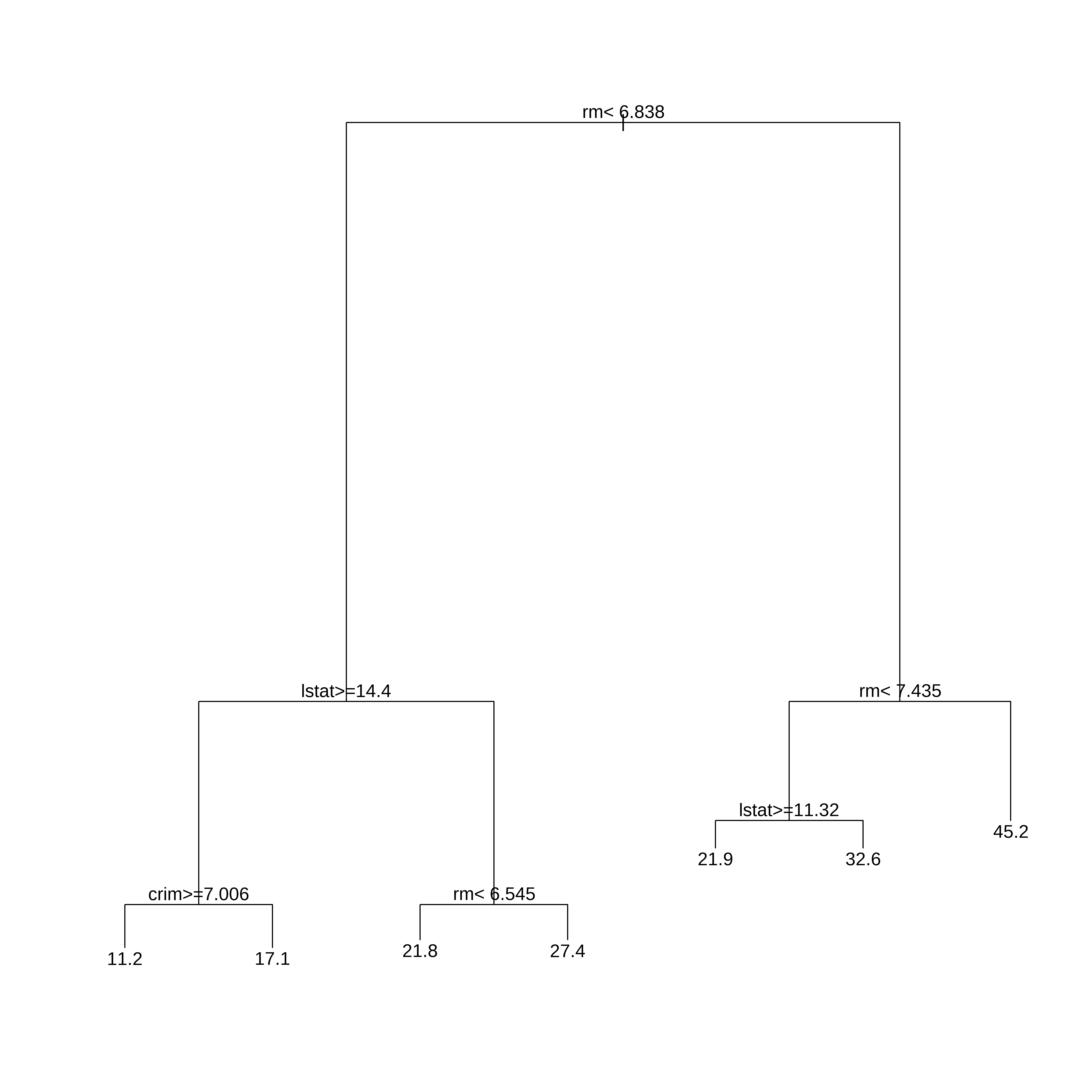
Gráfico de importancia das variáveis
A importancia das variáveis é calculada com base nos resultados das melhores partições
# Gráfico de Importância de variável
var_imp <- varImp(arvreg2)
var_imp$importance %>%
rownames_to_column(var = "Variável") %>%
ggplot(aes(x = reorder(Variável, Overall), y = Overall)) +
geom_col(fill = "steelblue") +
coord_flip() +
labs(title = "Importância das Variáveis na Árvore de Regressão",
x = "Variável",
y = "Importância (Overall)") +
theme_minimal()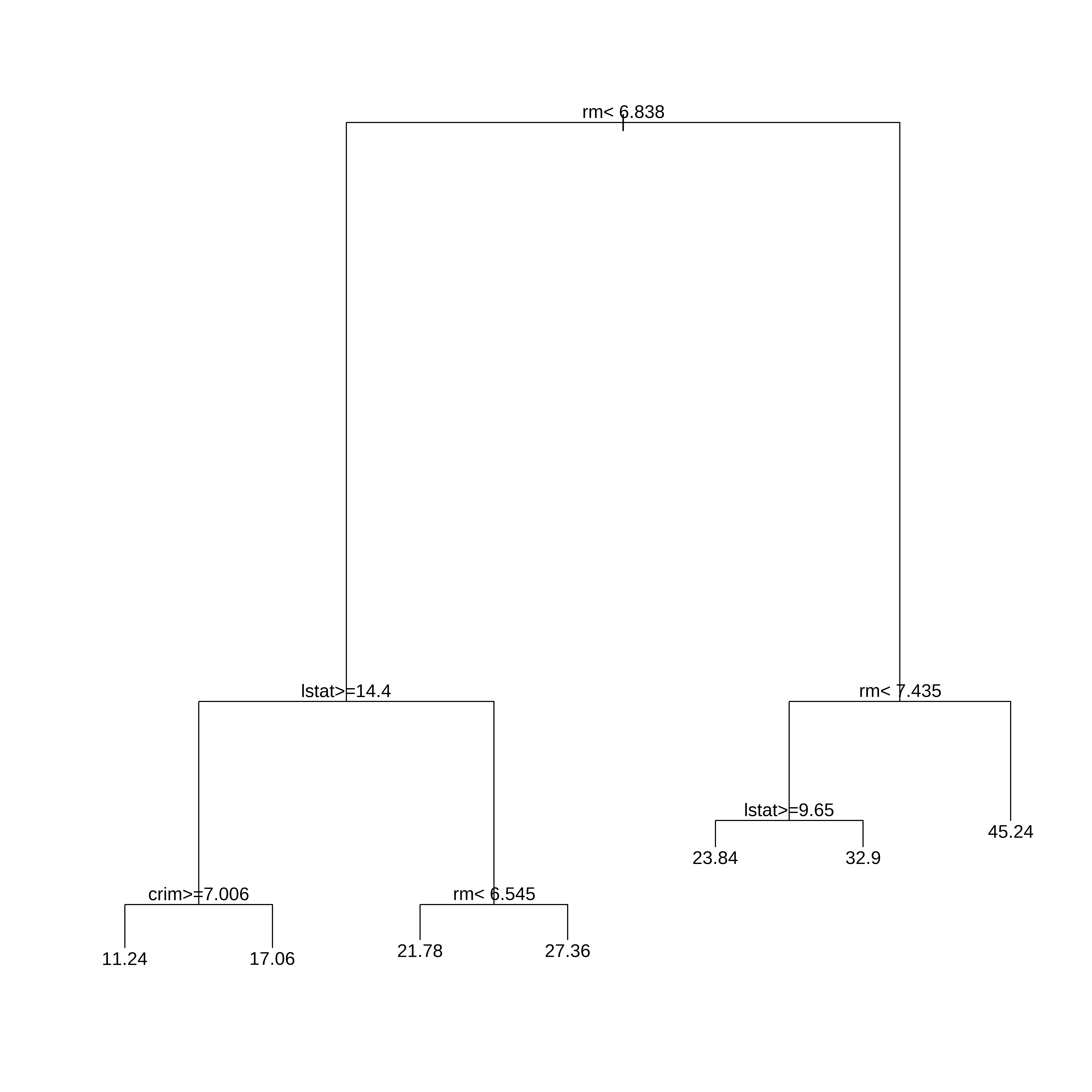
Previsões
# Regras de Decisão
arvreg2$finalModel#> n= 381
#>
#> node), split, n, deviance, yval
#> * denotes terminal node
#>
#> 1) root 381 29517.3100 22.24934
#> 2) rm< 7.0105 337 14500.6500 20.17745
#> 4) lstat>=14.4 133 2641.8500 14.88271
#> 8) nox>=0.607 84 1154.7830 12.88571
#> 16) lstat>=19.645 45 393.3764 10.63111 *
#> 17) lstat< 19.645 39 268.7236 15.48718 *
#> 9) nox< 0.607 49 577.8082 18.30612 *
#> 5) lstat< 14.4 204 5699.3640 23.62941
#> 10) rm< 6.543 156 3036.0710 22.16090
#> 20) dis>=1.68515 149 1307.6110 21.81141 *
#> 21) dis< 1.68515 7 1322.8800 29.60000 *
#> 11) rm>=6.543 48 1233.5100 28.40208
#> 22) lstat>=5.195 29 213.9524 26.04828 *
#> 23) lstat< 5.195 19 613.6495 31.99474 *
#> 3) rm>=7.0105 44 2489.9250 38.11818
#> 6) rm< 7.435 26 382.4815 32.73846 *
#> 7) rm>=7.435 18 268.0578 45.88889 *#> 1 3 4 5 9 19
#> 31.99474 32.73846 31.99474 32.73846 18.30612 21.81141# Calcula os erros de previsão
RMSE(previsao2, conj_teste$medv)#> [1] 4.882214Vamos comparar com Regressão Multipla
library(leaps)
## Cria uma função de predição para o leaps
predict.regsubsets <- function(object,newdata,id,...){
form <- as.formula(object$call[[2]])
mat <- model.matrix(form,newdata)
coefi <- coef(object,id=id)
mat[,names(coefi)]%*%coefi
}
set.seed(2025)
envelopes <- sample(rep(1:5,length=nrow(conj_treino)))
table(envelopes)#> envelopes
#> 1 2 3 4 5
#> 77 76 76 76 76erro_cv <- matrix(NA,5,13)
for(k in 1:5){
melh_ajus <- regsubsets(medv ~ ., data=conj_treino[envelopes!=k,],
nvmax=13,method="forward")
for(i in 1:13){
prev <- predict(melh_ajus, conj_treino[envelopes==k,],id=i)
erro_cv[k,i] <- mean( (conj_treino$medv[envelopes==k]-prev)^2)
}
}
rmse_cv <- sqrt(apply(erro_cv,2,mean)) # Erro medio quadratico de cada modelo
plot(rmse_cv,pch=19,type="b") 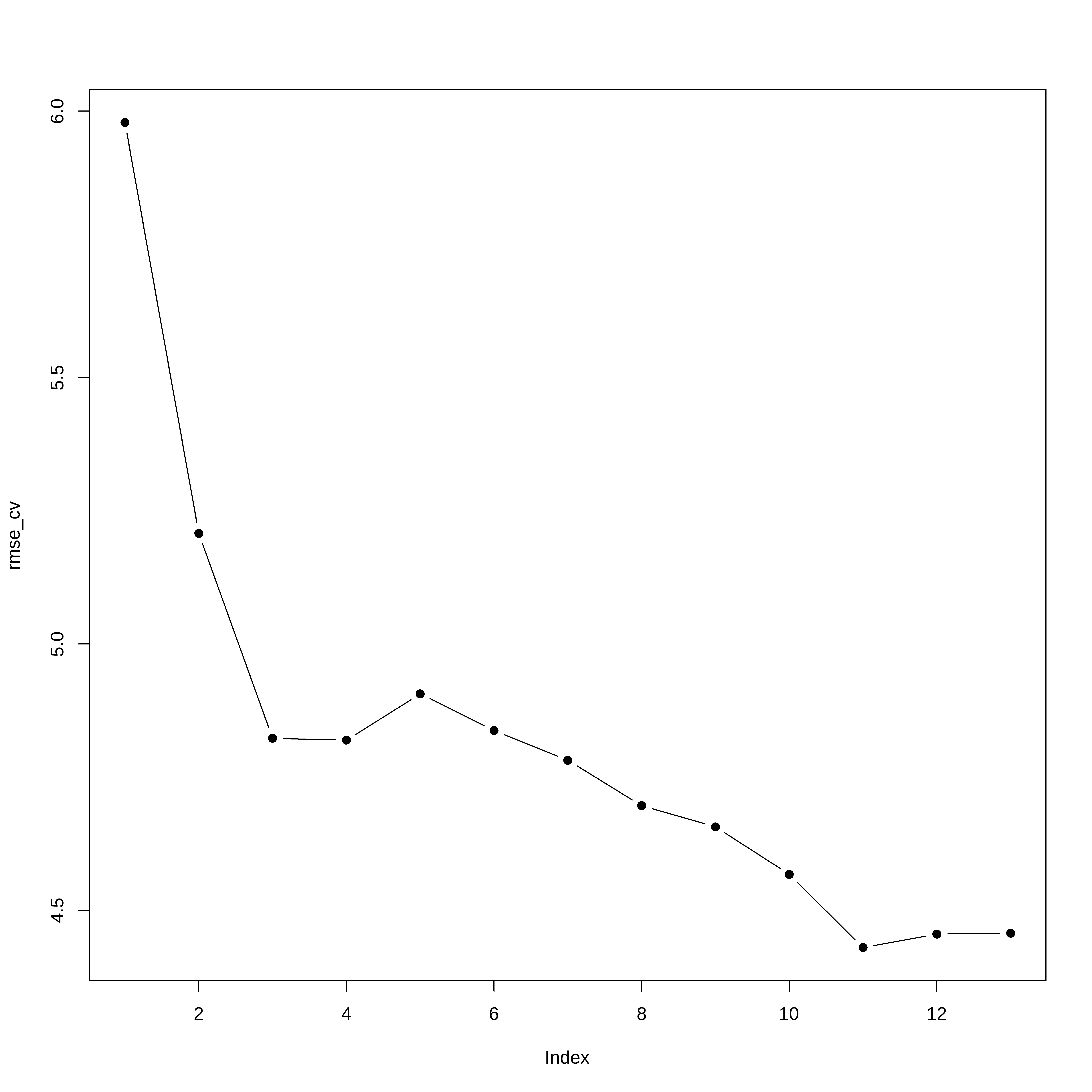
Obtem a fórmula do modelo
coef(melh_ajus, 11)#> (Intercept) crim zn chas nox
#> 34.693940504 -0.124555425 0.050468728 4.160779593 -21.520063917
#> rm dis rad tax ptratio
#> 4.179728433 -1.594068072 0.357136658 -0.013259547 -0.837573459
#> black lstat
#> 0.008077009 -0.500287649Teste com o conjunto de teste
E se usarmos outra semente?
## Vamos criar os conjuntos de treino teste e desenvolver a arvore
## com todas as variáveis.
set.seed(23)
indice <- createDataPartition(dados$medv, times=1, p=0.75, list=FALSE)
conj_treino <- dados[indice,]
arvreg1s <- rpart(medv ~ .,
data=conj_treino,
method="anova", #para arvore de regressão
control=rpart.control(minsplit=30,cp=0.01))
arvreg1s#> n= 381
#>
#> node), split, n, deviance, yval
#> * denotes terminal node
#>
#> 1) root 381 31257.8400 22.41496
#> 2) rm< 6.92 321 11786.7500 19.74361
#> 4) lstat>=14.4 133 2558.5280 14.88722
#> 8) crim>=6.99237 59 866.3051 11.96949 *
#> 9) crim< 6.99237 74 789.4865 17.21351 *
#> 5) lstat< 14.4 188 3872.3890 23.17926
#> 10) lstat>=5.51 165 2166.8920 22.28970
#> 20) lstat>=9.675 86 527.0414 20.76977 *
#> 21) lstat< 9.675 79 1224.8950 23.94430 *
#> 11) lstat< 5.51 23 638.2548 29.56087 *
#> 3) rm>=6.92 60 4925.2370 36.70667
#> 6) rm< 7.437 39 1510.0630 31.43077
#> 12) ptratio>=18.55 10 750.6440 25.84000 *
#> 13) ptratio< 18.55 29 339.0703 33.35862 *
#> 7) rm>=7.437 21 313.5495 46.50476 *## Colocar as duas arvores lado a lado
par(mfrow=c(1,2))
rpart.plot(arvreg1s)
rpart.plot(arvreg2$finalModel)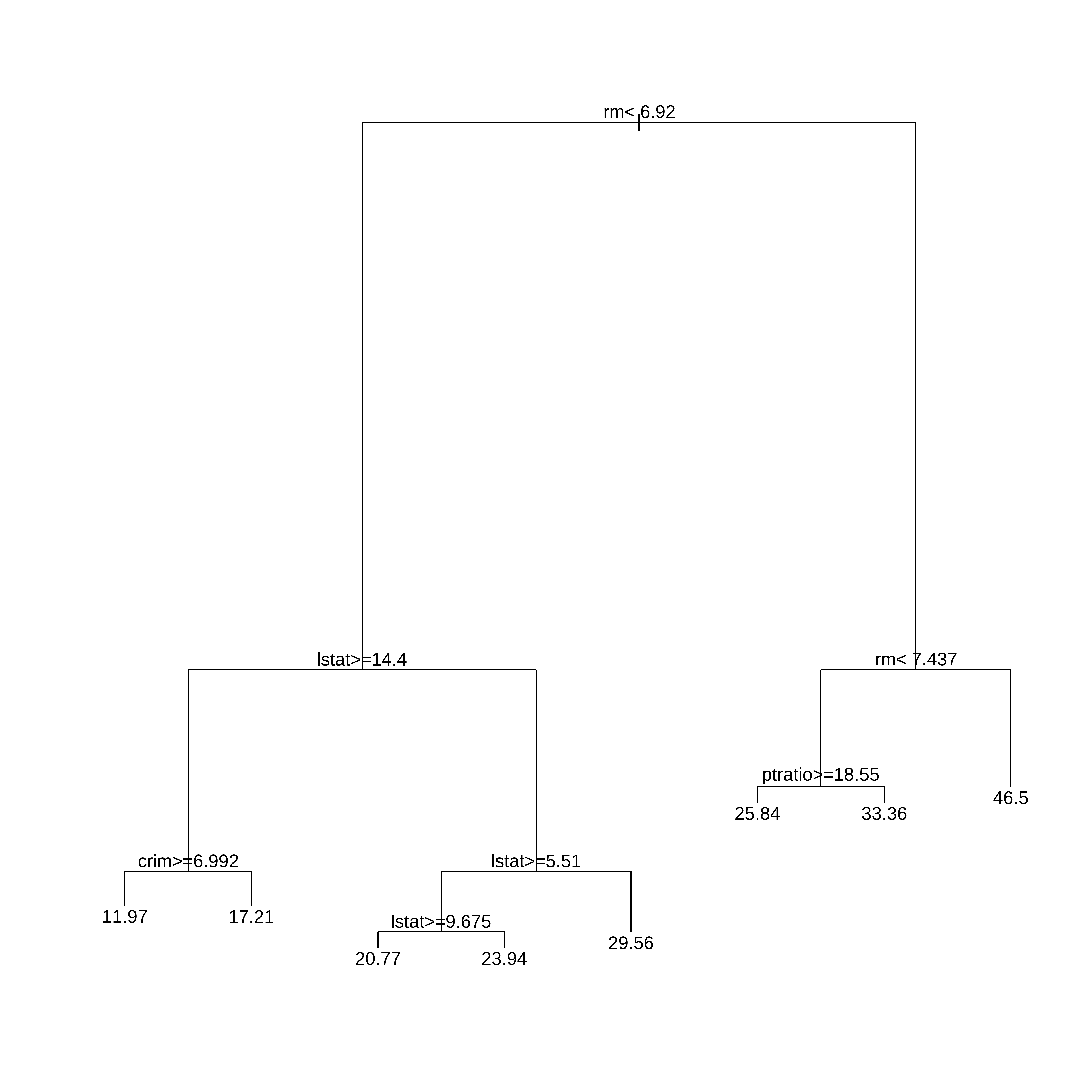
Esta é a principal fragilidade da árvore (única), qualquer mudança na amostra pode trazer uma configuração diferente. ## Fim