Arvores de Regressão - LIME e SHAP
Bibliotecas
Avaliando, selecionando dados
Treino e Teste com todas as variáveis
## Vamos criar os conjuntos de treino teste e desenvolver a arvore
## com todas as variáveis.
library(caret)
set.seed(21)
indice <- createDataPartition(dados$medv, times=1, p=0.75, list=FALSE)
conj_treino <- dados[indice,]
conj_teste <- dados[-indice,]
head(conj_treino) crim zn indus chas nox rm age dis rad tax ptratio black lstat
1 0.00632 18.0 2.31 0 0.538 6.575 65.2 4.0900 1 296 15.3 396.90 4.98
3 0.02729 0.0 7.07 0 0.469 7.185 61.1 4.9671 2 242 17.8 392.83 4.03
4 0.03237 0.0 2.18 0 0.458 6.998 45.8 6.0622 3 222 18.7 394.63 2.94
5 0.06905 0.0 2.18 0 0.458 7.147 54.2 6.0622 3 222 18.7 396.90 5.33
6 0.02985 0.0 2.18 0 0.458 6.430 58.7 6.0622 3 222 18.7 394.12 5.21
7 0.08829 12.5 7.87 0 0.524 6.012 66.6 5.5605 5 311 15.2 395.60 12.43
medv
1 24.0
3 34.7
4 33.4
5 36.2
6 28.7
7 22.9head(conj_teste) crim zn indus chas nox rm age dis rad tax ptratio black lstat
2 0.02731 0.0 7.07 0 0.469 6.421 78.9 4.9671 2 242 17.8 396.90 9.14
10 0.17004 12.5 7.87 0 0.524 6.004 85.9 6.5921 5 311 15.2 386.71 17.10
12 0.11747 12.5 7.87 0 0.524 6.009 82.9 6.2267 5 311 15.2 396.90 13.27
16 0.62739 0.0 8.14 0 0.538 5.834 56.5 4.4986 4 307 21.0 395.62 8.47
19 0.80271 0.0 8.14 0 0.538 5.456 36.6 3.7965 4 307 21.0 288.99 11.69
23 1.23247 0.0 8.14 0 0.538 6.142 91.7 3.9769 4 307 21.0 396.90 18.72
medv
2 21.6
10 18.9
12 18.9
16 19.9
19 20.2
23 15.2Treinamento do modelo Random Forest
modelo_rf <- ranger(
formula = medv ~ .,
data = conj_treino,
num.trees = 500
)# Wrapper para predições
model_type.ranger <- function(x, ...) {
"regression"
}
predict_model.ranger <- function(x, newdata, ...) {
data.frame(Response = predict(x, data = newdata)$predictions)
}
# Criando explicador LIME
explainer_lime <- lime(
x = conj_treino[, setdiff(names(conj_treino), "medv")],
model = modelo_rf
)
# Explicação para a primeira observação do teste
explanation_lime <- lime::explain(
x = conj_teste[1, setdiff(names(conj_teste),"medv"), drop=FALSE],
explainer = explainer_lime,
n_features = 5
)
# Gráfico LIME
lime::plot_features(explanation_lime)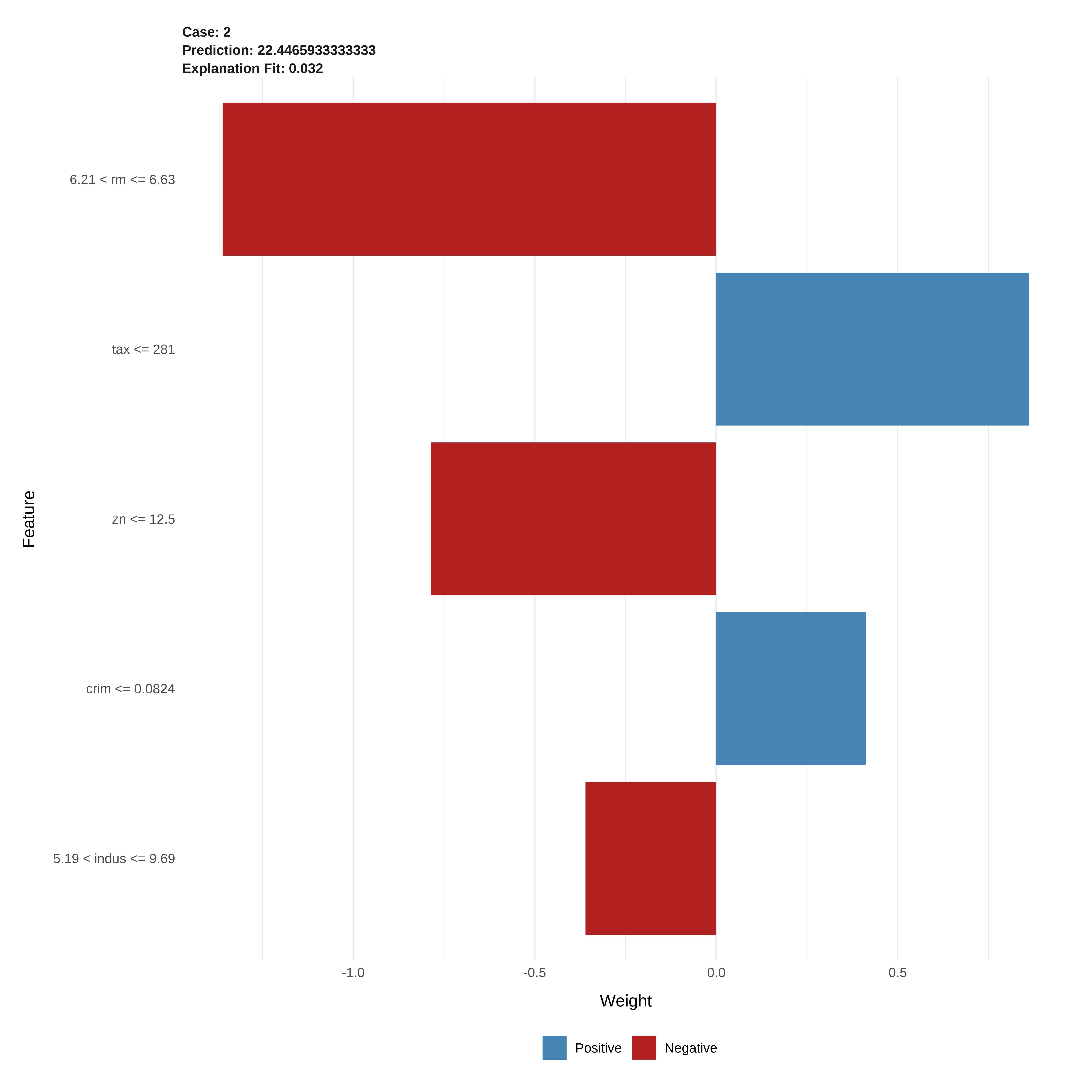
# Criando objeto predictor para o iml
predictor_shap <- Predictor$new(
model = modelo_rf,
data = conj_treino[, -which(names(conj_treino) == "medv")],
y = conj_treino$medv
)
# SHAP para a mesma observação
shap <- Shapley$new(predictor_shap, x.interest = conj_teste[1, -which(names(conj_teste) == "medv")])
# Gráfico SHAP
plot(shap)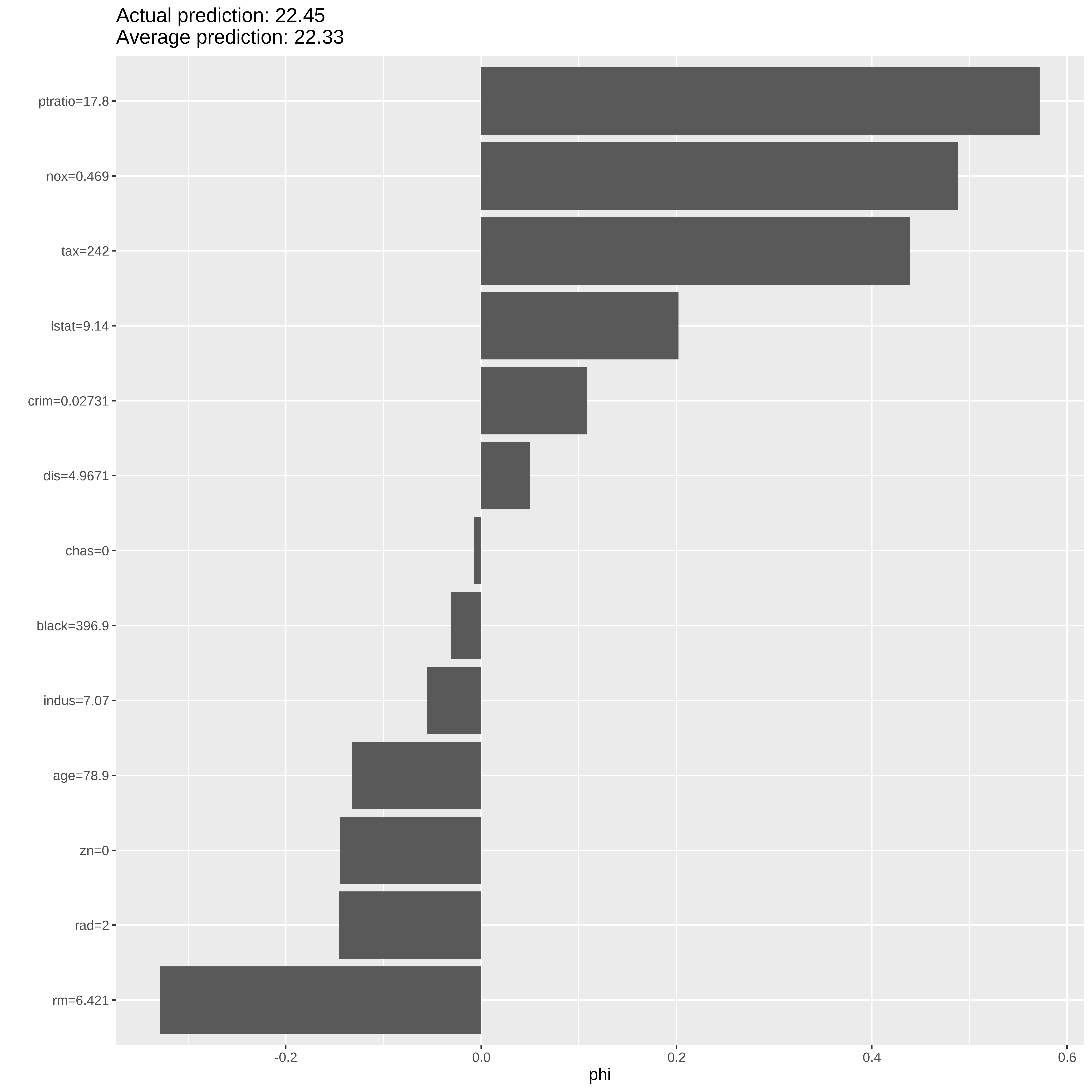
# Gráfico SHAP com shapviz
# Matriz de preditores
X <- conj_treino[, setdiff(names(conj_treino), "medv")]
# Função de predição para fastshap
pred_fun <- function(object, newdata) {
predict(object, data = newdata)$predictions
}
# Calculando valores SHAP com fastshap
shap_values <- fastshap::explain(
object = modelo_rf,
X = X,
pred_wrapper = pred_fun,
nsim = 100 # número de permutações (pode ajustar)
)
# Criando objeto shapviz
sv_rf <- shapviz(shap_values, X = X)
# Visualizações
sv_importance(sv_rf, kind="bee")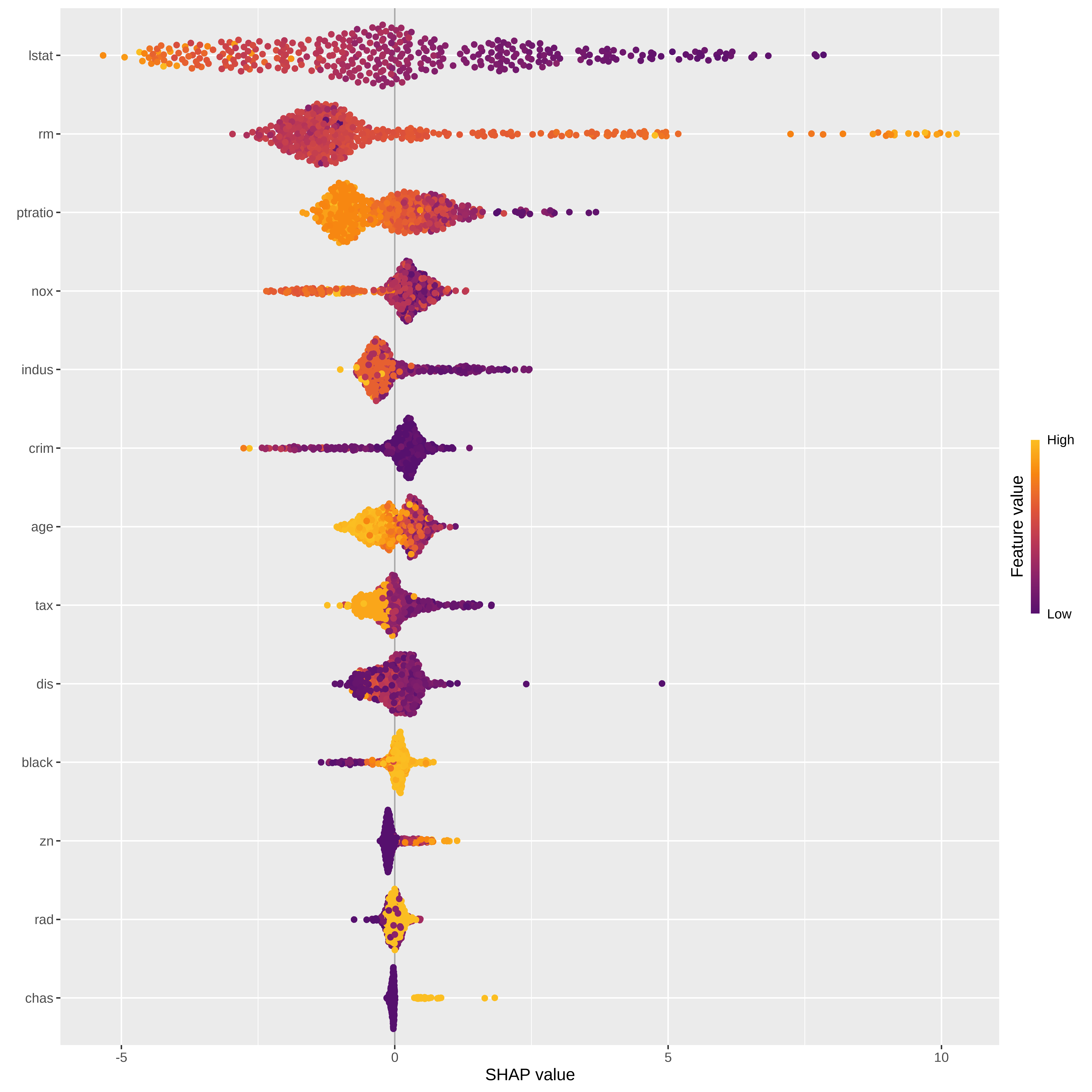
sv_dependence(sv_rf, v = "lstat")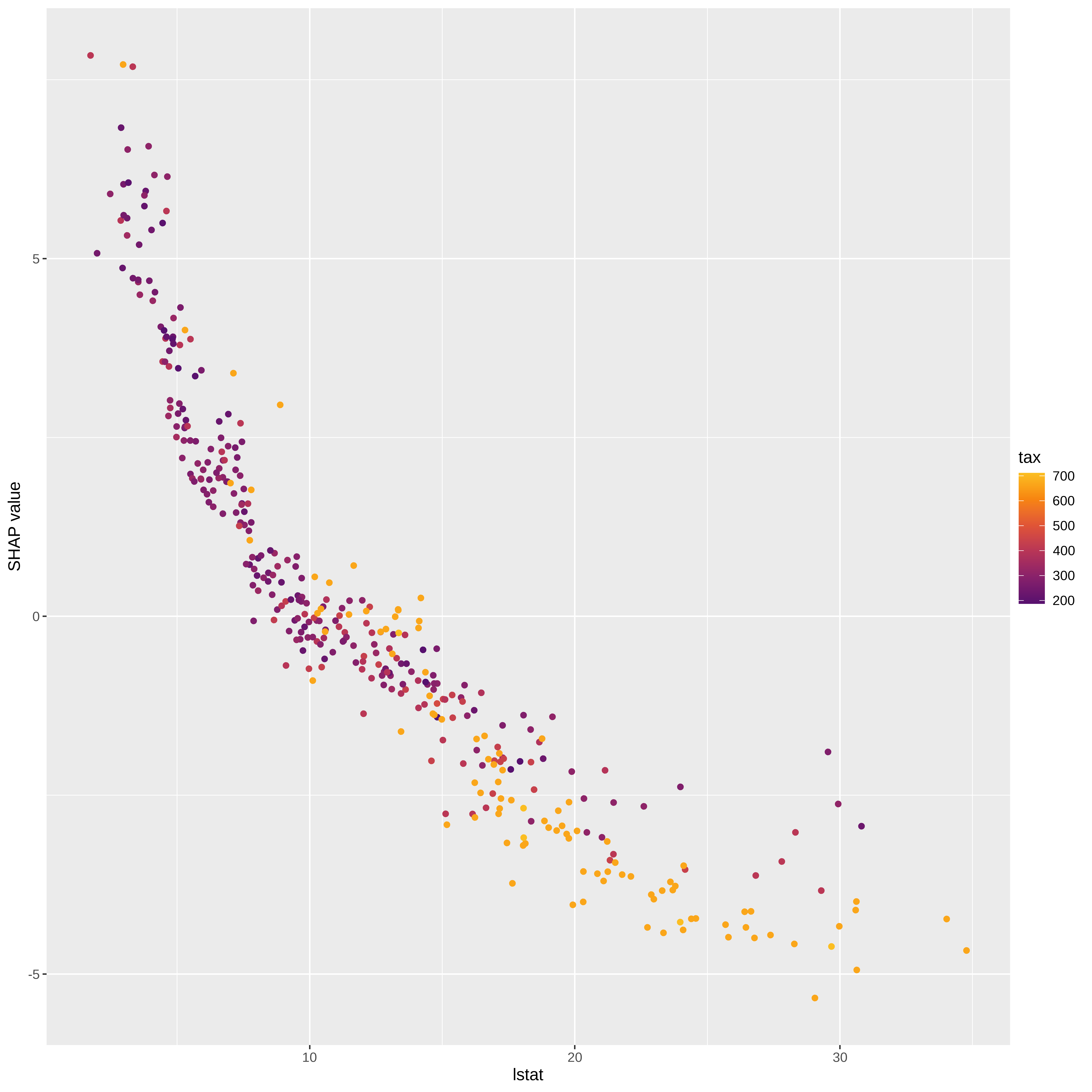
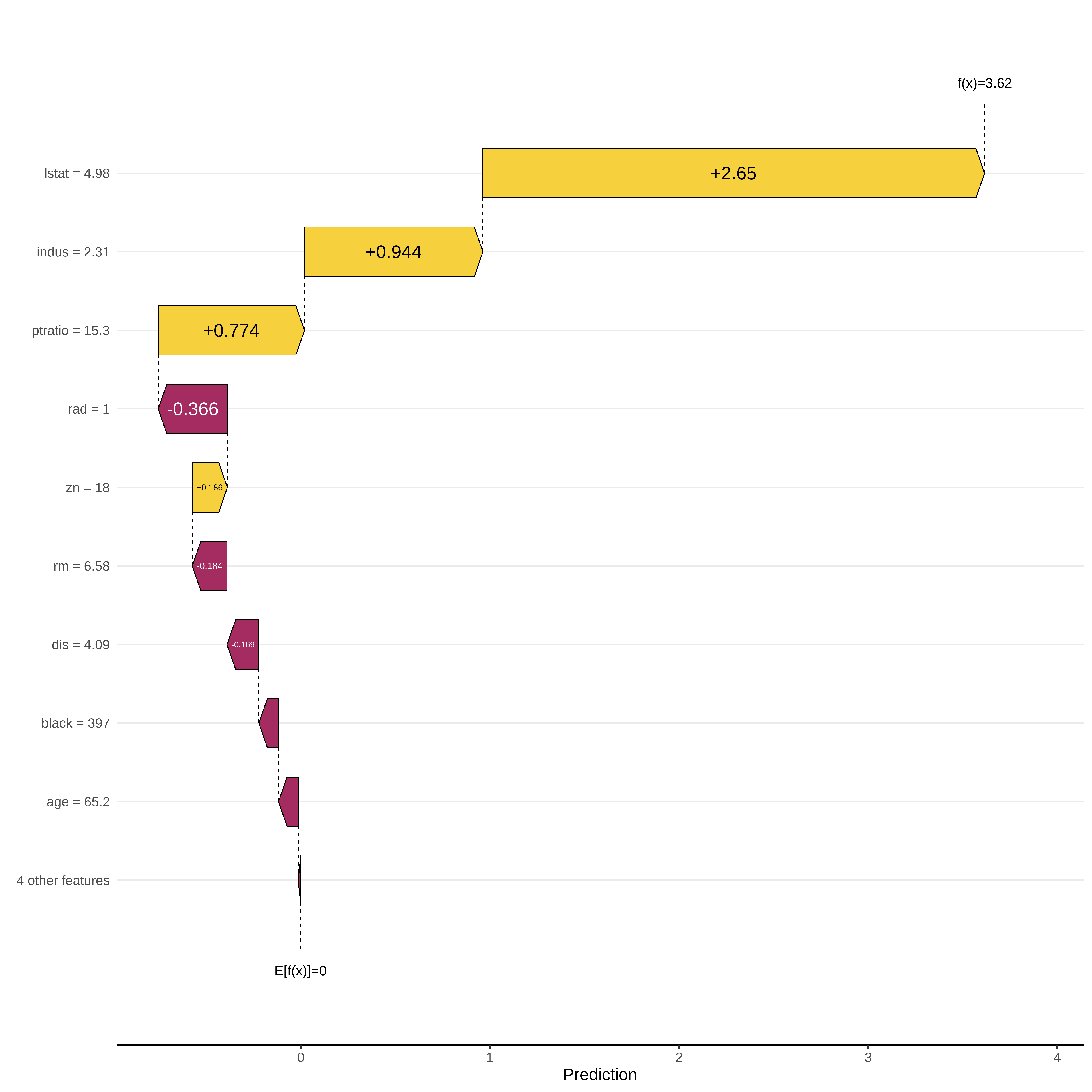
sv_waterfall(sv_rf, row_id = 1)