Classificação - Primeiros Passos
Primeiros Passos
Carregando Bibliotecas
Traduzindo
head(heights)#> sex height
#> 1 Male 75
#> 2 Male 70
#> 3 Male 68
#> 4 Male 74
#> 5 Male 61
#> 6 Female 65str(heights)#> 'data.frame': 1050 obs. of 2 variables:
#> $ sex : Factor w/ 2 levels "Female","Male": 2 2 2 2 2 1 1 1 1 2 ...
#> $ height: num 75 70 68 74 61 65 66 62 66 67 ...alturas <- heights %>%
# traduz dados para o português e transforma polegada para metro
mutate(
sex = case_when(
sex == "Male" ~ "Masculino",
sex == "Female" ~ "Feminino"
),
altura = height * 2.54 / 100 ) %>%
rename( sexo = sex) %>%
select(sexo , altura)
head(alturas)#> sexo altura
#> 1 Masculino 1.9050
#> 2 Masculino 1.7780
#> 3 Masculino 1.7272
#> 4 Masculino 1.8796
#> 5 Masculino 1.5494
#> 6 Feminino 1.6510str(alturas)#> 'data.frame': 1050 obs. of 2 variables:
#> $ sexo : chr "Masculino" "Masculino" "Masculino" "Masculino" ...
#> $ altura: num 1.9 1.78 1.73 1.88 1.55 ...Alterando
alturas <- data.frame(sexo = factor(alturas$sexo), altura = alturas$altura)
str(alturas)#> 'data.frame': 1050 obs. of 2 variables:
#> $ sexo : Factor w/ 2 levels "Feminino","Masculino": 2 2 2 2 2 1 1 1 1 2 ...
#> $ altura: num 1.9 1.78 1.73 1.88 1.55 ...head(alturas)#> sexo altura
#> 1 Masculino 1.9050
#> 2 Masculino 1.7780
#> 3 Masculino 1.7272
#> 4 Masculino 1.8796
#> 5 Masculino 1.5494
#> 6 Feminino 1.6510Definindo x e y
y <- alturas$sexo
x <- alturas$alturaTreino e Teste
set.seed(1234)
indice_teste <- createDataPartition(y, times = 1, p = 0.2, list = FALSE)
conj_teste <- alturas[indice_teste, ]
conj_treino <- alturas[-indice_teste, ]
head(conj_teste)#> sexo altura
#> 4 Masculino 1.8796
#> 7 Feminino 1.6764
#> 9 Feminino 1.6764
#> 14 Masculino 1.7272
#> 16 Masculino 1.6764
#> 25 Masculino 1.8288str(conj_teste)#> 'data.frame': 211 obs. of 2 variables:
#> $ sexo : Factor w/ 2 levels "Feminino","Masculino": 2 1 1 2 2 2 2 2 2 2 ...
#> $ altura: num 1.88 1.68 1.68 1.73 1.68 ...summary(conj_teste)#> sexo altura
#> Feminino : 48 Min. :1.366
#> Masculino:163 1st Qu.:1.676
#> Median :1.727
#> Mean :1.735
#> 3rd Qu.:1.803
#> Max. :2.032head(conj_treino)#> sexo altura
#> 1 Masculino 1.9050
#> 2 Masculino 1.7780
#> 3 Masculino 1.7272
#> 5 Masculino 1.5494
#> 6 Feminino 1.6510
#> 8 Feminino 1.5748str(conj_treino)#> 'data.frame': 839 obs. of 2 variables:
#> $ sexo : Factor w/ 2 levels "Feminino","Masculino": 2 2 2 2 1 1 2 2 2 2 ...
#> $ altura: num 1.9 1.78 1.73 1.55 1.65 ...summary(conj_treino)#> sexo altura
#> Feminino :190 Min. :1.270
#> Masculino:649 1st Qu.:1.676
#> Median :1.750
#> Mean :1.736
#> 3rd Qu.:1.803
#> Max. :2.100Chutar a resposta!!!!!
Aqui vamos aletoriamente chutar a resposta.
Calcula precisão
mean(y_chapeu == conj_teste$sexo)#> [1] 0.549763Melhorando
Agora vamos tentar melhorar o chute inicial usando as alturas das pessoas.
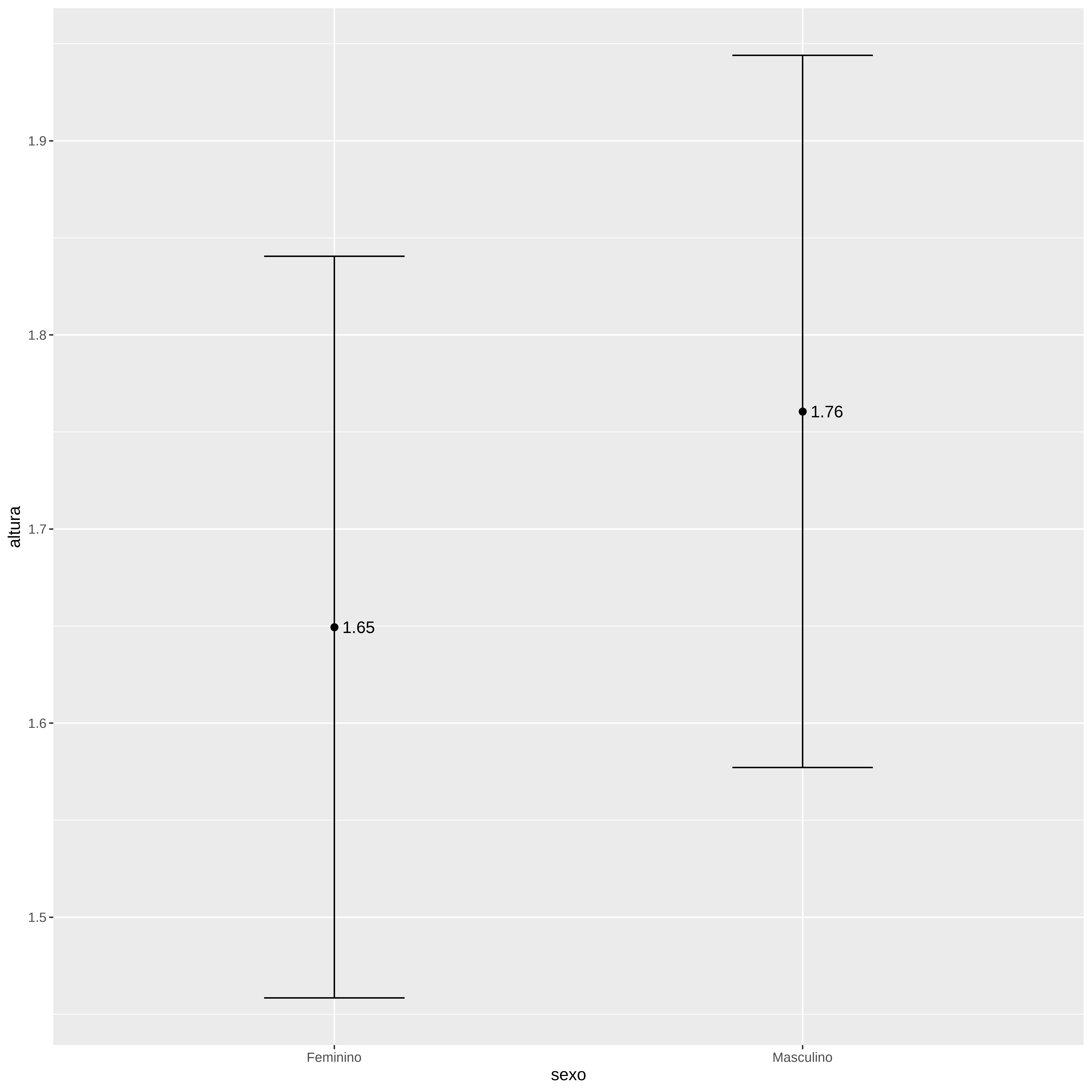
Primeiro vamos visualizar a distribuição das alturas entre homens e mulheres
library(ggplot2)
ggplot(alturas, aes(x = sexo, y = altura)) +
stat_summary(
geom = "errorbar",
fun.max = function(y) mean(y) + 2*sd(y),
fun.min = function(y) mean(y) - 2*sd(y),
width = .3
) +
stat_summary(
geom = "point",
fun = mean,
size = 2
) +
stat_summary(
geom = "text",
fun = mean,
aes(label = after_stat(round(y, 2))),
hjust = -0.3
)
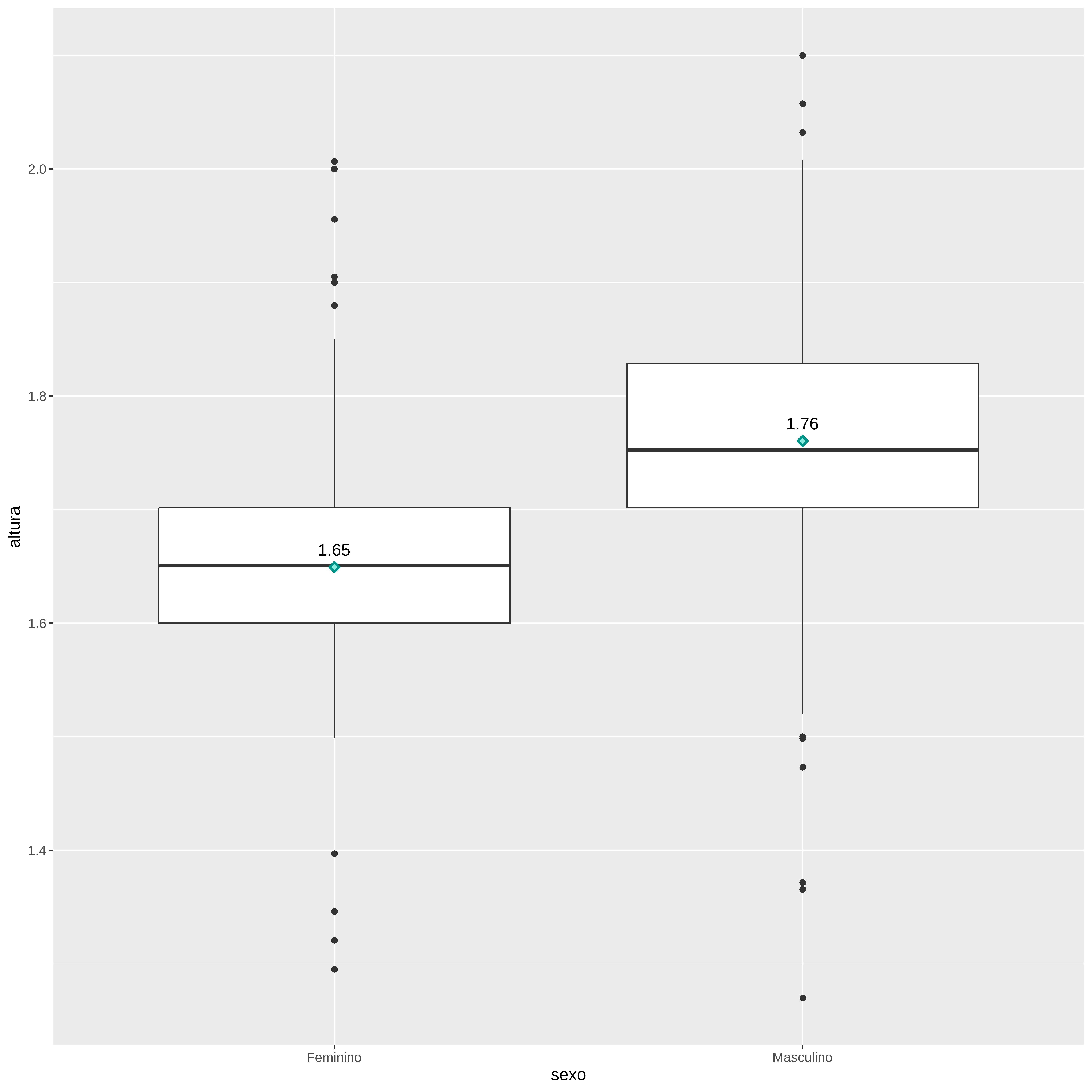
ggplot(alturas, aes(x = sexo, y = altura)) +
geom_boxplot() +
stat_summary(
geom = "point",
fun = mean,
shape = 23,
color = "#00978A",
fill = "#88EBDD",
stroke = 1.2
) +
stat_summary(
geom = "text",
fun = mean,
aes(label = after_stat(round(y, 2))),
vjust = -1.0
)
#> # A tibble: 2 × 3
#> sexo `mean(altura)` `sd(altura)`
#> <fct> <dbl> <dbl>
#> 1 Feminino 1.65 0.0955
#> 2 Masculino 1.76 0.0917media_Homem <- mean(alturas[alturas$sexo=="Masculino",]$altura)
desv_pad_Homem <- sd(alturas[alturas$sexo=="Masculino",]$altura)
media_Homem#> [1] 1.760595desv_pad_Homem#> [1] 0.09172media_Homem - 2*desv_pad_Homem#> [1] 1.577155y_chapeu <- ifelse(x > media_Homem - 2*desv_pad_Homem , "Masculino", "Feminino") %>% factor(levels = levels(conj_teste$sexo))
mean(y == y_chapeu)#> [1] 0.7933333Qual o melhor ponto de corte?
pontodecorte <- seq(1.54, 1.78, by = 0.01)
precisao <- map_dbl(pontodecorte, function(x){
y_chapeu <- ifelse(conj_treino$altura > x, "Masculino", "Feminino") %>%
factor(levels = levels(conj_teste$sexo))
mean(y_chapeu == conj_treino$sexo)
})
max(precisao)#> [1] 0.8307509melhor_ponto <- pontodecorte[which.max(precisao)]
melhor_ponto#> [1] 1.65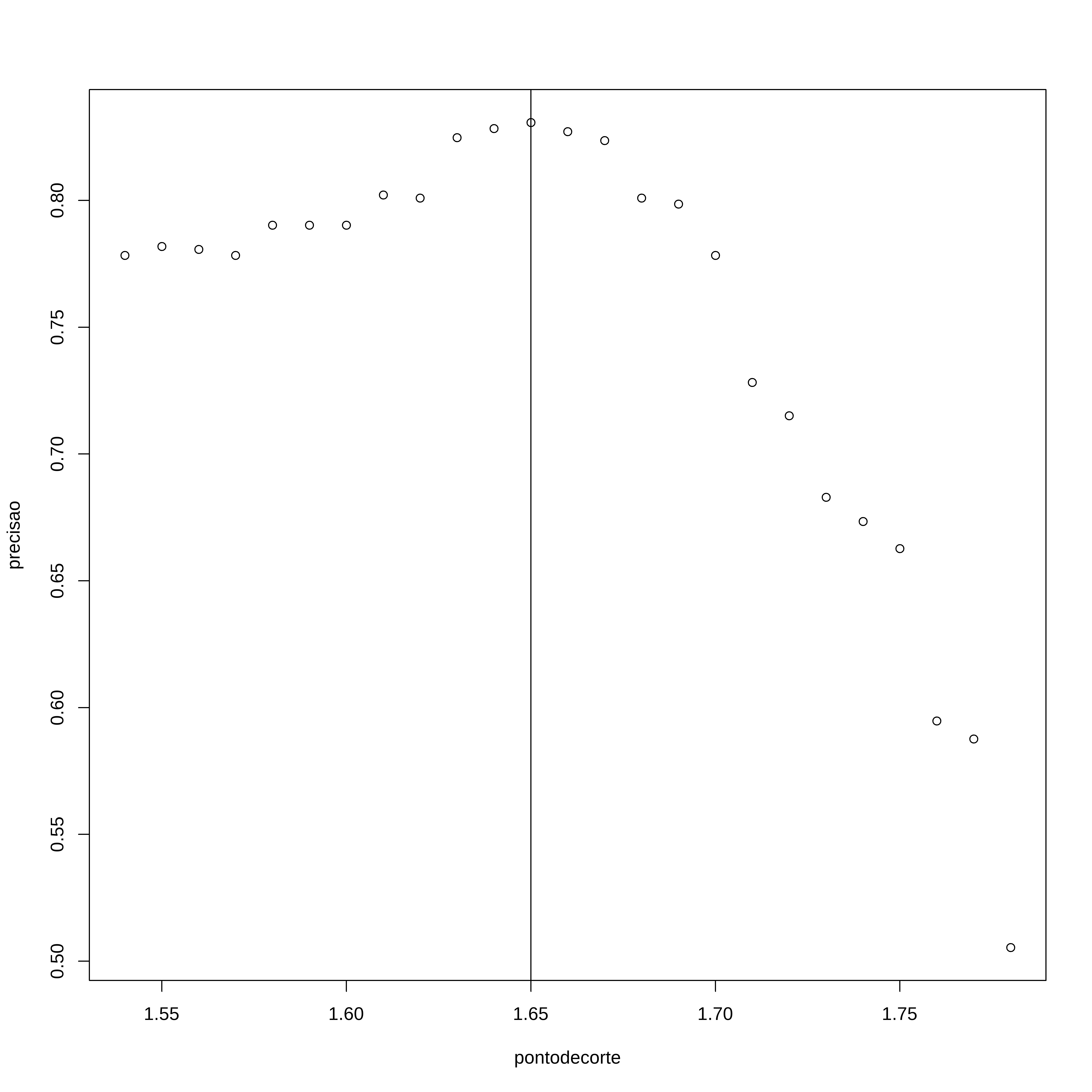
y_chapeu <- ifelse(conj_teste$altura > melhor_ponto, "Masculino", "Feminino") %>%
factor(levels = levels(conj_teste$sexo))
y_chapeu <- factor(y_chapeu)
mean(y_chapeu == conj_teste$sexo)#> [1] 0.8530806Melhorando o gráfico
library(ggplot2)
dados <- data.frame(pontodecorte, precisao)
ggplot(data=dados, aes(x=pontodecorte, y=precisao)) + geom_point() +
geom_vline(xintercept = melhor_ponto, linetype="dashed", color="red")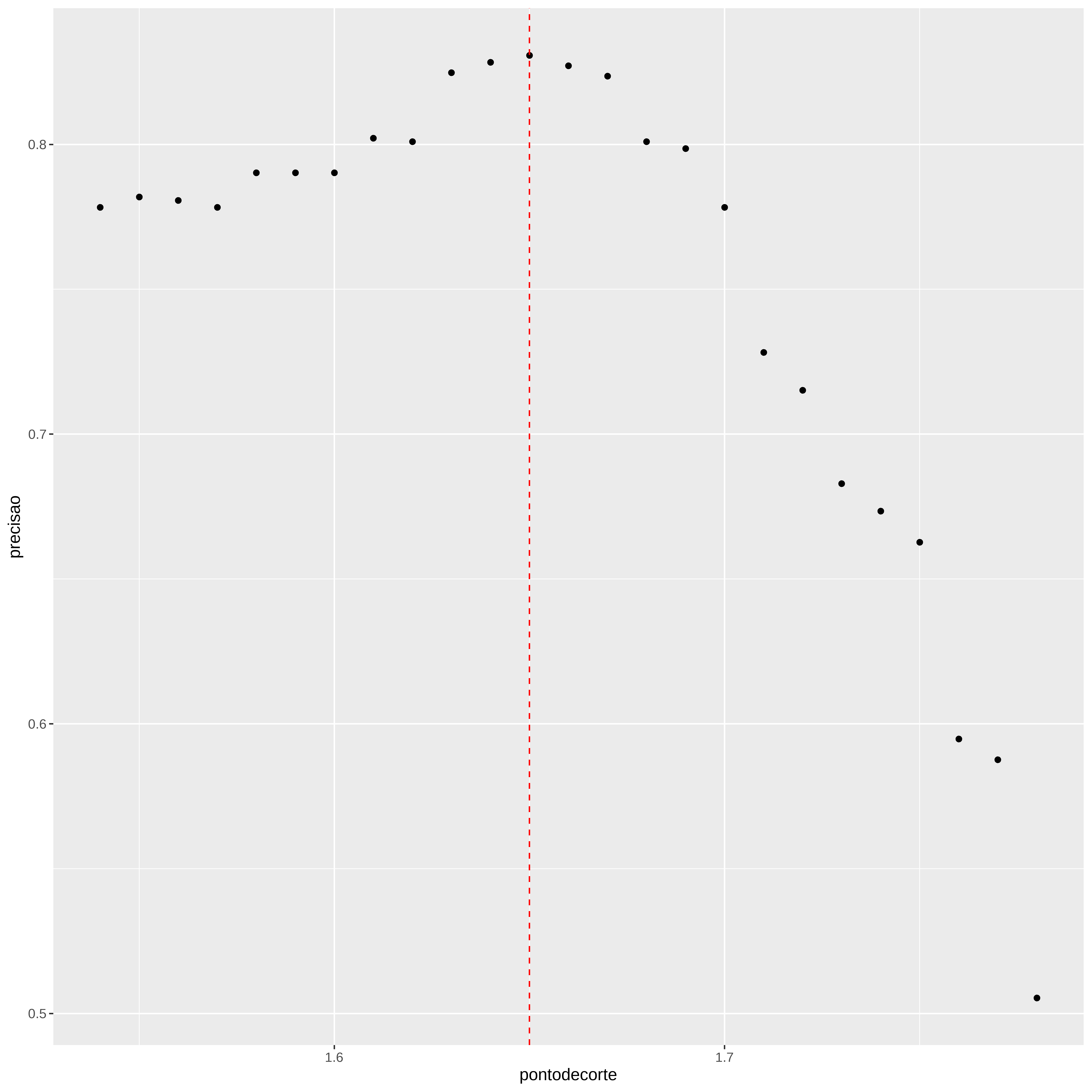
Matriz de Confusão
table(previsto = y_chapeu, real = conj_teste$sexo)#> real
#> previsto Feminino Masculino
#> Feminino 26 9
#> Masculino 22 154conj_teste %>%
mutate(y_chapeu = y_chapeu) %>%
group_by(sexo) %>%
summarize(precisao = mean(y_chapeu == sexo))#> # A tibble: 2 × 2
#> sexo precisao
#> <fct> <dbl>
#> 1 Feminino 0.542
#> 2 Masculino 0.945confusionMatrix(data = y_chapeu, reference = conj_teste$sexo)#> Confusion Matrix and Statistics
#>
#> Reference
#> Prediction Feminino Masculino
#> Feminino 26 9
#> Masculino 22 154
#>
#> Accuracy : 0.8531
#> 95% CI : (0.798, 0.8979)
#> No Information Rate : 0.7725
#> P-Value [Acc > NIR] : 0.002359
#>
#> Kappa : 0.5378
#>
#> Mcnemar's Test P-Value : 0.031141
#>
#> Sensitivity : 0.5417
#> Specificity : 0.9448
#> Pos Pred Value : 0.7429
#> Neg Pred Value : 0.8750
#> Prevalence : 0.2275
#> Detection Rate : 0.1232
#> Detection Prevalence : 0.1659
#> Balanced Accuracy : 0.7432
#>
#> 'Positive' Class : Feminino
#> Maximizando a estatística F1
pontodecorte <- seq(1.54, 1.78, by = 0.01)
F_1 <- map_dbl(pontodecorte, function(x){
y_chapeu <- ifelse(conj_treino$altura > x, "Masculino", "Feminino") %>%
factor(levels = levels(conj_teste$sexo))
F_meas(data = y_chapeu, reference = factor(conj_treino$sexo))
})
max(F_1)#> [1] 0.616092melhor_ponto <- pontodecorte[which.max(F_1)]
melhor_ponto#> [1] 1.68dados <- data.frame(pontodecorte, F_1)
ggplot(data=dados, aes(x=pontodecorte, y=F_1)) + geom_point() +
geom_vline(xintercept = melhor_ponto, linetype="dashed", color="red")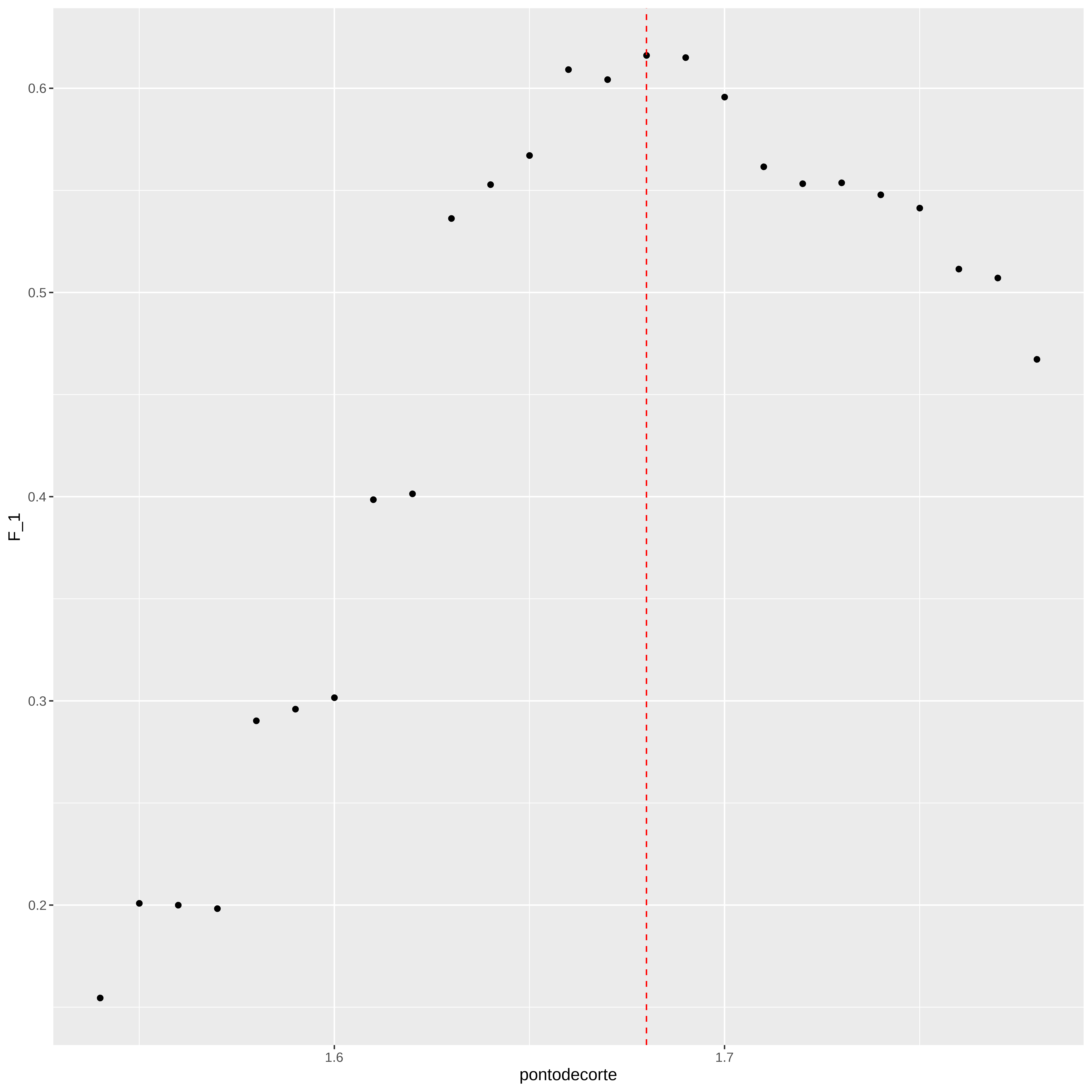
y_chapeu <- ifelse(conj_teste$altura > melhor_ponto, "Masculino", "Feminino") %>%
factor(levels = levels(conj_teste$sexo))
confusionMatrix(data = y_chapeu, reference = conj_teste$sexo)#> Confusion Matrix and Statistics
#>
#> Reference
#> Prediction Feminino Masculino
#> Feminino 32 26
#> Masculino 16 137
#>
#> Accuracy : 0.8009
#> 95% CI : (0.7406, 0.8526)
#> No Information Rate : 0.7725
#> P-Value [Acc > NIR] : 0.1839
#>
#> Kappa : 0.4724
#>
#> Mcnemar's Test P-Value : 0.1649
#>
#> Sensitivity : 0.6667
#> Specificity : 0.8405
#> Pos Pred Value : 0.5517
#> Neg Pred Value : 0.8954
#> Prevalence : 0.2275
#> Detection Rate : 0.1517
#> Detection Prevalence : 0.2749
#> Balanced Accuracy : 0.7536
#>
#> 'Positive' Class : Feminino
#> Voltando a chutar
Curva ROC
ggplot() +
geom_abline(intercept = 0, slope = 1.) +
labs(x = "Taxa Positiva Falsa (1 - Especificidade)",
y = "Taxa Positiva Verdadeira (Sensibilidade)",
title = "Curva ROC") +
scale_x_continuous(limits = c(0, 1.0)) +
scale_y_continuous(limits = c(0, 1.0))
Testando probabilidades
probs <- seq(0, 1, length.out = 10)
chutando <- map_df(probs, function(p){
y_chapeu <-
sample(c("Masculino", "Feminino"), length(indice_teste), replace = TRUE, prob=c(p, 1-p)) %>% factor(levels = levels(conj_teste$sexo))
list(metodo = "Chutando",
TPF = 1 - specificity(y_chapeu, conj_teste$sexo),
TPV = sensitivity(y_chapeu, conj_teste$sexo))
})
chutando %>%
ggplot(aes(TPF, TPV)) +
geom_line() +
geom_point() +
geom_abline(intercept = 0, slope = 1.) +
labs(x = "Taxa Positiva Falsa (1 - Especificidade)",
y = "Taxa Positiva Verdadeira (Sensibilidade)",
title = "Curva ROC Método Chutando") +
scale_x_continuous(limits = c(0, 1.0)) +
scale_y_continuous(limits = c(0, 1.0))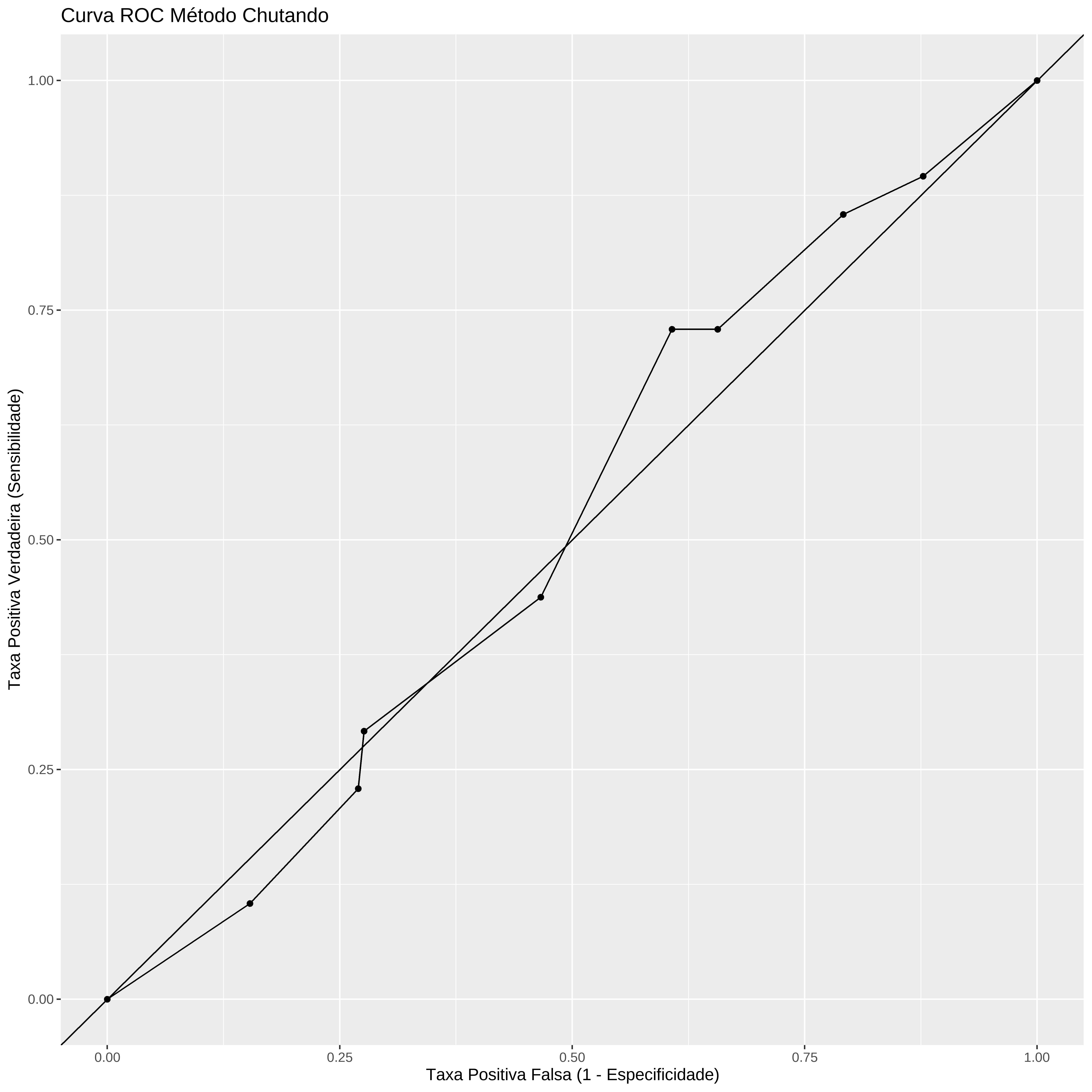
Comparando os dois métodos
pontodecorte <- seq(1.27 , 1.92, by = 0.01)
altura_ptdecorte <- map_df(pontodecorte, function(x){
y_chapeu <- ifelse(conj_teste$altura > x, "Masculino", "Feminino") %>%
factor(levels = levels(conj_teste$sexo))
list(metodo = "Corte por Altura",
TPF = 1 - specificity(y_chapeu, conj_teste$sexo),
TPV = sensitivity(y_chapeu, conj_teste$sexo))
})
bind_rows(chutando, altura_ptdecorte) %>%
ggplot(aes(TPF, TPV, color = metodo)) +
geom_line() +
geom_point() +
geom_abline(intercept = 0, slope = 1.) +
labs(x = "Taxa Positiva Falsa (1 - Especificidade)",
y = "Taxa Positiva Verdadeira (Sensibilidade)",
title = "Curva ROC") +
scale_x_continuous(limits = c(0, 1.0)) +
scale_y_continuous(limits = c(0, 1.0))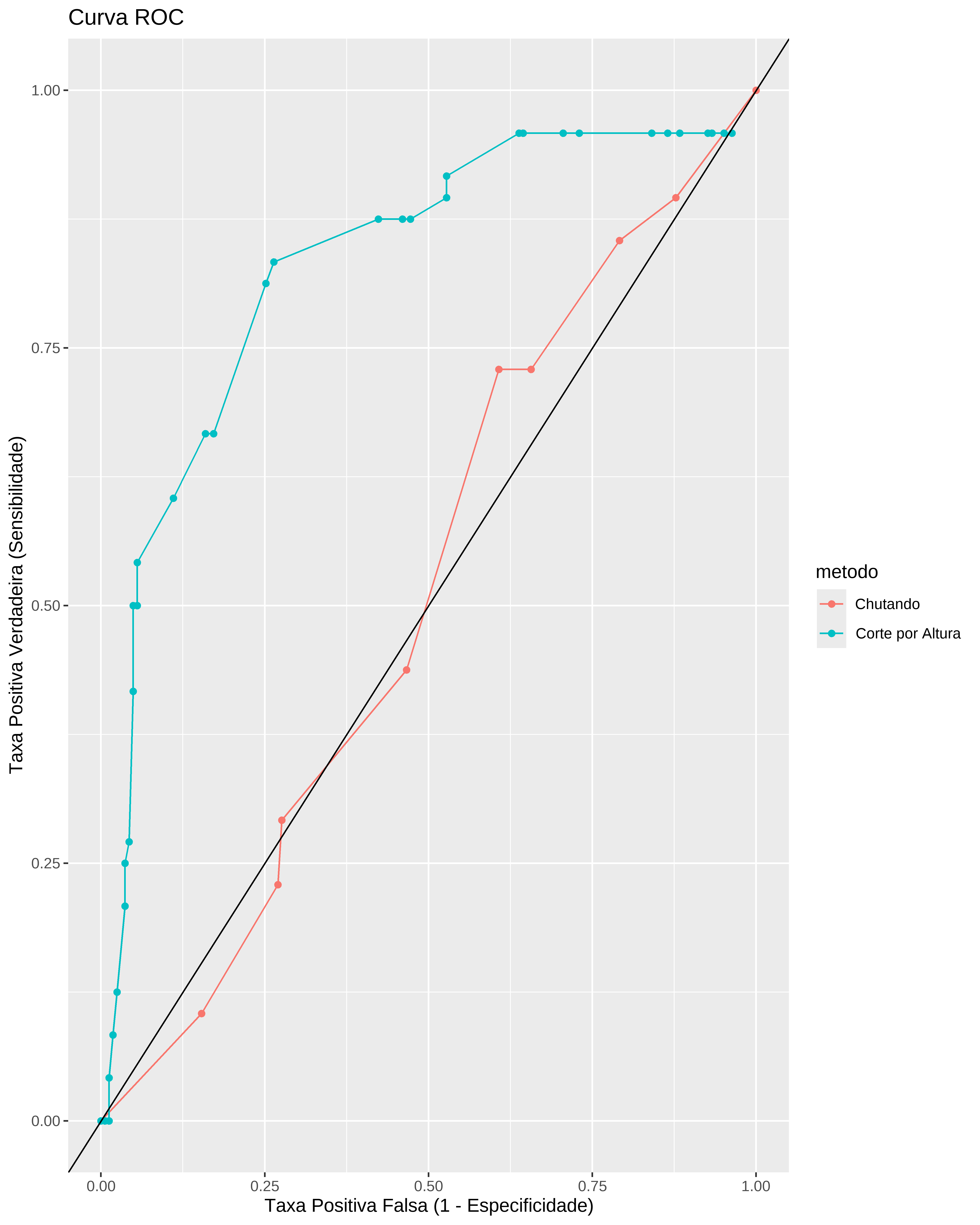
Pontos da curva ROC
map_df(pontodecorte, function(x){
y_chapeu <- ifelse(conj_teste$altura > x, "Masculino", "Feminino") %>%
factor(levels = c("Masculino", "Feminino"))
list(metodo = "Corte por Altura",
corte = round(x, 2),
TPF = 1 - specificity(y_chapeu, conj_teste$sexo),
TPV = sensitivity(y_chapeu, conj_teste$sexo))
}) %>%
ggplot(aes(TPF, TPV, label = corte)) +
geom_line() +
geom_point() +
geom_text(nudge_y = - 0.05, size = 3, check_overlap = TRUE) +
scale_x_continuous(limits = c(0, 1.0)) +
scale_y_continuous(limits = c(0, 1.0))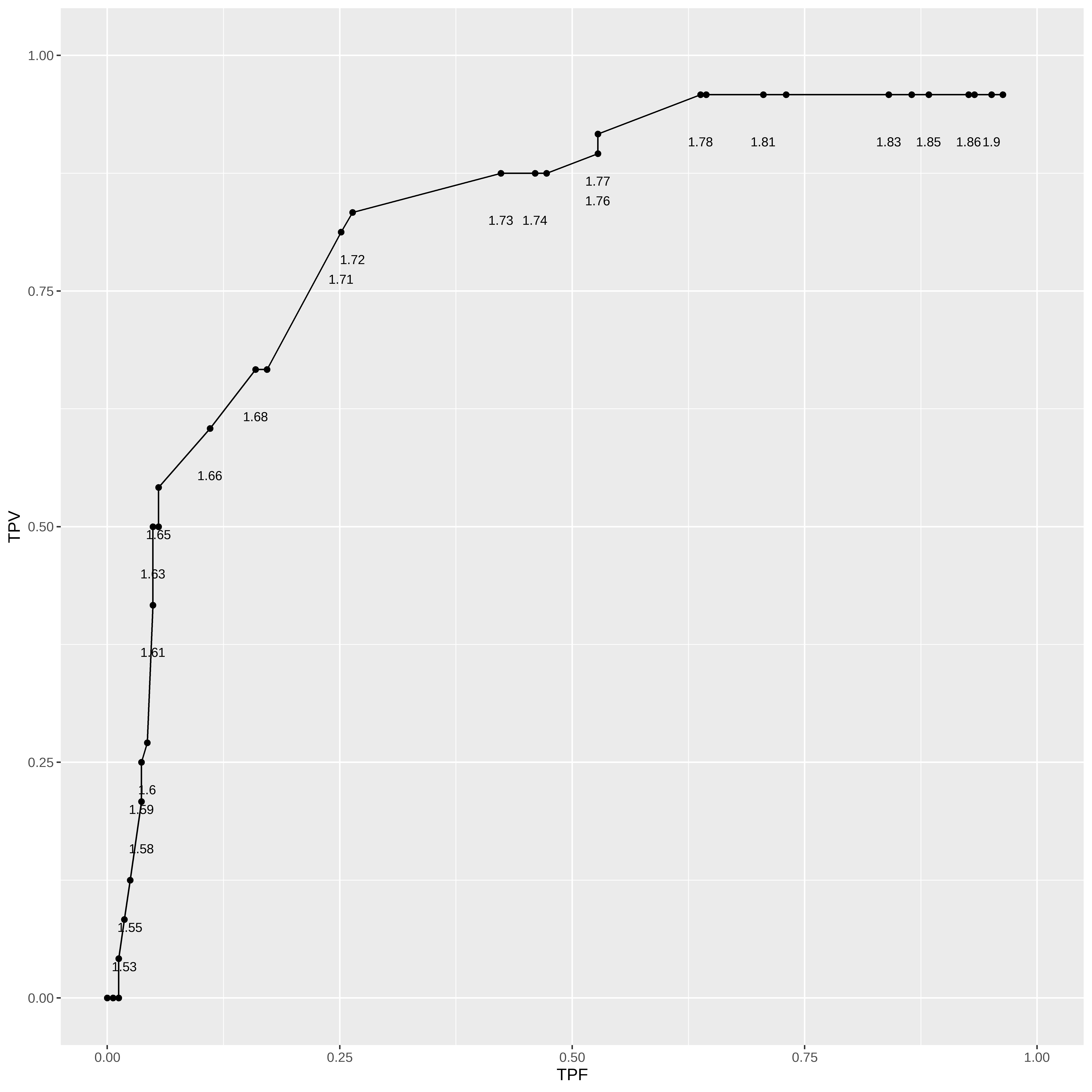
Bayes Ingênuo (Naive Bayes)
params <- conj_treino %>%
group_by(sexo) %>%
summarize(media = mean(altura), desvpad = sd(altura))
params#> # A tibble: 2 × 3
#> sexo media desvpad
#> <fct> <dbl> <dbl>
#> 1 Feminino 1.65 0.0947
#> 2 Masculino 1.76 0.0925#> [1] 0.2264601x <- conj_teste$altura
f1 <- dnorm(x, params$media[1], params$desvpad[1])
f0 <- dnorm(x, params$media[2], params$desvpad[2])
p_chapeu_bayes <- f1*pi / (f1*pi + f0*(1 - pi))
y_chapeu_bayes <- ifelse(p_chapeu_bayes > 0.5, "Feminino", "Masculino")
confusionMatrix(factor(y_chapeu_bayes), conj_teste$sexo, positive="Feminino")#> Confusion Matrix and Statistics
#>
#> Reference
#> Prediction Feminino Masculino
#> Feminino 20 7
#> Masculino 28 156
#>
#> Accuracy : 0.8341
#> 95% CI : (0.7769, 0.8817)
#> No Information Rate : 0.7725
#> P-Value [Acc > NIR] : 0.0174801
#>
#> Kappa : 0.4419
#>
#> Mcnemar's Test P-Value : 0.0007232
#>
#> Sensitivity : 0.41667
#> Specificity : 0.95706
#> Pos Pred Value : 0.74074
#> Neg Pred Value : 0.84783
#> Prevalence : 0.22749
#> Detection Rate : 0.09479
#> Detection Prevalence : 0.12796
#> Balanced Accuracy : 0.68686
#>
#> 'Positive' Class : Feminino
#>