Arvores de Classificação - GBM
Bibliotecas
Dados
Vamos começar a aplicar a metodologia de árvores usando árvores de classificação para analisar os dados existentes em Carseats. Este conjunto de dados (simulado) é sobre venda de assentos de criança para carros. Ele tem 400 observações das seguintes variáveis (11), cujos nomes serão convertidos para o português:
Sales: vendas em unidades (em mil) em cada local
CompPrice: preço cobrado pelo competidor em cada local
Income: nível de renda da comunidade local (em mil US$)
Advertising: orçamento local de propaganda (em mil US$)
Population: população na região (em mil)
Price: preço cobrado pela empresa em cada local
ShelveLoc: um fator com níveis Ruim, Bom e Medio indicando a qualidade da localização das prateleiras para os assentos em cada lugar
Age: idade media da população local
Education: nível de educação em cada local
Urban: um fator Sim e Não indicando se a loja esta em uma área urbana ou rural
US: um fator indicando se a loja é nos EUA ou não
Neste dados, Sales é a variável resposta, só que ela é uma variável contínua, por este motivo vamos usá-la para criar uma variável binária. Vamos usar a função ifelse() para criar a variável binária, que chamaremos de alta, ela assume os valores Sim se Sales for maior que 8 e assume o valor Não caso contrário:
Sales CompPrice Income Advertising
Min. : 0.000 Min. : 77 Min. : 21.00 Min. : 0.000
1st Qu.: 5.390 1st Qu.:115 1st Qu.: 42.75 1st Qu.: 0.000
Median : 7.490 Median :125 Median : 69.00 Median : 5.000
Mean : 7.496 Mean :125 Mean : 68.66 Mean : 6.635
3rd Qu.: 9.320 3rd Qu.:135 3rd Qu.: 91.00 3rd Qu.:12.000
Max. :16.270 Max. :175 Max. :120.00 Max. :29.000
Population Price ShelveLoc Age Education
Min. : 10.0 Min. : 24.0 Bad : 96 Min. :25.00 Min. :10.0
1st Qu.:139.0 1st Qu.:100.0 Good : 85 1st Qu.:39.75 1st Qu.:12.0
Median :272.0 Median :117.0 Medium:219 Median :54.50 Median :14.0
Mean :264.8 Mean :115.8 Mean :53.32 Mean :13.9
3rd Qu.:398.5 3rd Qu.:131.0 3rd Qu.:66.00 3rd Qu.:16.0
Max. :509.0 Max. :191.0 Max. :80.00 Max. :18.0
Urban US
No :118 No :142
Yes:282 Yes:258
str(Carseats)'data.frame': 400 obs. of 11 variables:
$ Sales : num 9.5 11.22 10.06 7.4 4.15 ...
$ CompPrice : num 138 111 113 117 141 124 115 136 132 132 ...
$ Income : num 73 48 35 100 64 113 105 81 110 113 ...
$ Advertising: num 11 16 10 4 3 13 0 15 0 0 ...
$ Population : num 276 260 269 466 340 501 45 425 108 131 ...
$ Price : num 120 83 80 97 128 72 108 120 124 124 ...
$ ShelveLoc : Factor w/ 3 levels "Bad","Good","Medium": 1 2 3 3 1 1 3 2 3 3 ...
$ Age : num 42 65 59 55 38 78 71 67 76 76 ...
$ Education : num 17 10 12 14 13 16 15 10 10 17 ...
$ Urban : Factor w/ 2 levels "No","Yes": 2 2 2 2 2 1 2 2 1 1 ...
$ US : Factor w/ 2 levels "No","Yes": 2 2 2 2 1 2 1 2 1 2 ...Manipulando os dados
cad_crianca <- Carseats %>% rename(vendas = Sales,
preco_comp = CompPrice,
renda = Income,
propaganda = Advertising,
populacao = Population,
preco = Price,
local_prat = ShelveLoc,
idade = Age,
educacao = Education,
urbano = Urban,
eua = US)
cad_crianca <- cad_crianca %>%
mutate(vendaAlta = ifelse(vendas > 8, "Alta", "Baixa")) %>%
mutate(vendaAlta = as.factor(vendaAlta)) %>%
select(-vendas) # Remover Sales original
# Verificar distribuição
table(cad_crianca$vendaAlta)
Alta Baixa
164 236 str(cad_crianca)'data.frame': 400 obs. of 11 variables:
$ preco_comp: num 138 111 113 117 141 124 115 136 132 132 ...
$ renda : num 73 48 35 100 64 113 105 81 110 113 ...
$ propaganda: num 11 16 10 4 3 13 0 15 0 0 ...
$ populacao : num 276 260 269 466 340 501 45 425 108 131 ...
$ preco : num 120 83 80 97 128 72 108 120 124 124 ...
$ local_prat: Factor w/ 3 levels "Bad","Good","Medium": 1 2 3 3 1 1 3 2 3 3 ...
$ idade : num 42 65 59 55 38 78 71 67 76 76 ...
$ educacao : num 17 10 12 14 13 16 15 10 10 17 ...
$ urbano : Factor w/ 2 levels "No","Yes": 2 2 2 2 2 1 2 2 1 1 ...
$ eua : Factor w/ 2 levels "No","Yes": 2 2 2 2 1 2 1 2 1 2 ...
$ vendaAlta : Factor w/ 2 levels "Alta","Baixa": 1 1 1 2 2 1 2 1 2 2 ...summary(cad_crianca) preco_comp renda propaganda populacao
Min. : 77 Min. : 21.00 Min. : 0.000 Min. : 10.0
1st Qu.:115 1st Qu.: 42.75 1st Qu.: 0.000 1st Qu.:139.0
Median :125 Median : 69.00 Median : 5.000 Median :272.0
Mean :125 Mean : 68.66 Mean : 6.635 Mean :264.8
3rd Qu.:135 3rd Qu.: 91.00 3rd Qu.:12.000 3rd Qu.:398.5
Max. :175 Max. :120.00 Max. :29.000 Max. :509.0
preco local_prat idade educacao urbano
Min. : 24.0 Bad : 96 Min. :25.00 Min. :10.0 No :118
1st Qu.:100.0 Good : 85 1st Qu.:39.75 1st Qu.:12.0 Yes:282
Median :117.0 Medium:219 Median :54.50 Median :14.0
Mean :115.8 Mean :53.32 Mean :13.9
3rd Qu.:131.0 3rd Qu.:66.00 3rd Qu.:16.0
Max. :191.0 Max. :80.00 Max. :18.0
eua vendaAlta
No :142 Alta :164
Yes:258 Baixa:236
Treino e Teste
set.seed(21)
y <- cad_crianca$vendaAlta
indice_teste <- createDataPartition(y, times = 1, p = 0.2, list = FALSE)
conj_treino <- cad_crianca[-indice_teste,]
conj_teste <- cad_crianca[indice_teste,]
str(conj_treino)'data.frame': 319 obs. of 11 variables:
$ preco_comp: num 111 117 141 124 115 132 132 121 117 122 ...
$ renda : num 48 100 64 113 105 110 113 78 94 35 ...
$ propaganda: num 16 4 3 13 0 0 0 9 4 2 ...
$ populacao : num 260 466 340 501 45 108 131 150 503 393 ...
$ preco : num 83 97 128 72 108 124 124 100 94 136 ...
$ local_prat: Factor w/ 3 levels "Bad","Good","Medium": 2 3 1 1 3 3 3 1 2 3 ...
$ idade : num 65 55 38 78 71 76 76 26 50 62 ...
$ educacao : num 10 14 13 16 15 10 17 10 13 18 ...
$ urbano : Factor w/ 2 levels "No","Yes": 2 2 2 1 2 1 1 1 2 2 ...
$ eua : Factor w/ 2 levels "No","Yes": 2 2 1 2 1 1 2 2 2 1 ...
$ vendaAlta : Factor w/ 2 levels "Alta","Baixa": 1 2 2 1 2 2 2 1 1 2 ...prop.table(table(conj_treino$alta))numeric(0)str(conj_teste)'data.frame': 81 obs. of 11 variables:
$ preco_comp: num 138 113 136 125 139 98 115 131 157 118 ...
$ renda : num 73 35 81 90 32 118 54 84 53 71 ...
$ propaganda: num 11 10 15 2 0 0 0 11 0 4 ...
$ populacao : num 276 269 425 367 176 19 406 29 403 148 ...
$ preco : num 120 80 120 131 82 107 128 96 124 114 ...
$ local_prat: Factor w/ 3 levels "Bad","Good","Medium": 1 3 2 3 2 3 3 3 1 3 ...
$ idade : num 42 59 67 35 54 64 42 44 58 80 ...
$ educacao : num 17 12 10 18 11 17 17 17 16 13 ...
$ urbano : Factor w/ 2 levels "No","Yes": 2 2 2 2 1 2 2 1 2 2 ...
$ eua : Factor w/ 2 levels "No","Yes": 2 2 2 2 1 1 2 2 1 1 ...
$ vendaAlta : Factor w/ 2 levels "Alta","Baixa": 1 1 1 2 1 2 2 1 2 2 ...prop.table(table(conj_teste$alta))numeric(0)GBM
Ajustando um modelo GBM
set.seed(21)
# Definir grade de parâmetros
tune_grid <- expand.grid(
n.trees = c(100, 300, 500),
interaction.depth = c(1, 3, 5),
shrinkage = c(0.01, 0.1),
n.minobsinnode = c(5, 10)
)
# Treinamento com validação cruzada
ctrl <- trainControl(method = "cv", number = 5)
gbm_caret <- train(
vendaAlta ~ .,
data = conj_treino,
method = "gbm",
distribution = "bernoulli",
trControl = ctrl,
tuneGrid = tune_grid,
verbose = FALSE
)Melhor modelo
# Melhor modelo
gbm_caret$bestTune n.trees interaction.depth shrinkage n.minobsinnode
23 300 1 0.1 10Verificando os Resultados
# Previsões
pred_class <- predict(gbm_caret, newdata = conj_teste)
# Matriz de confusão
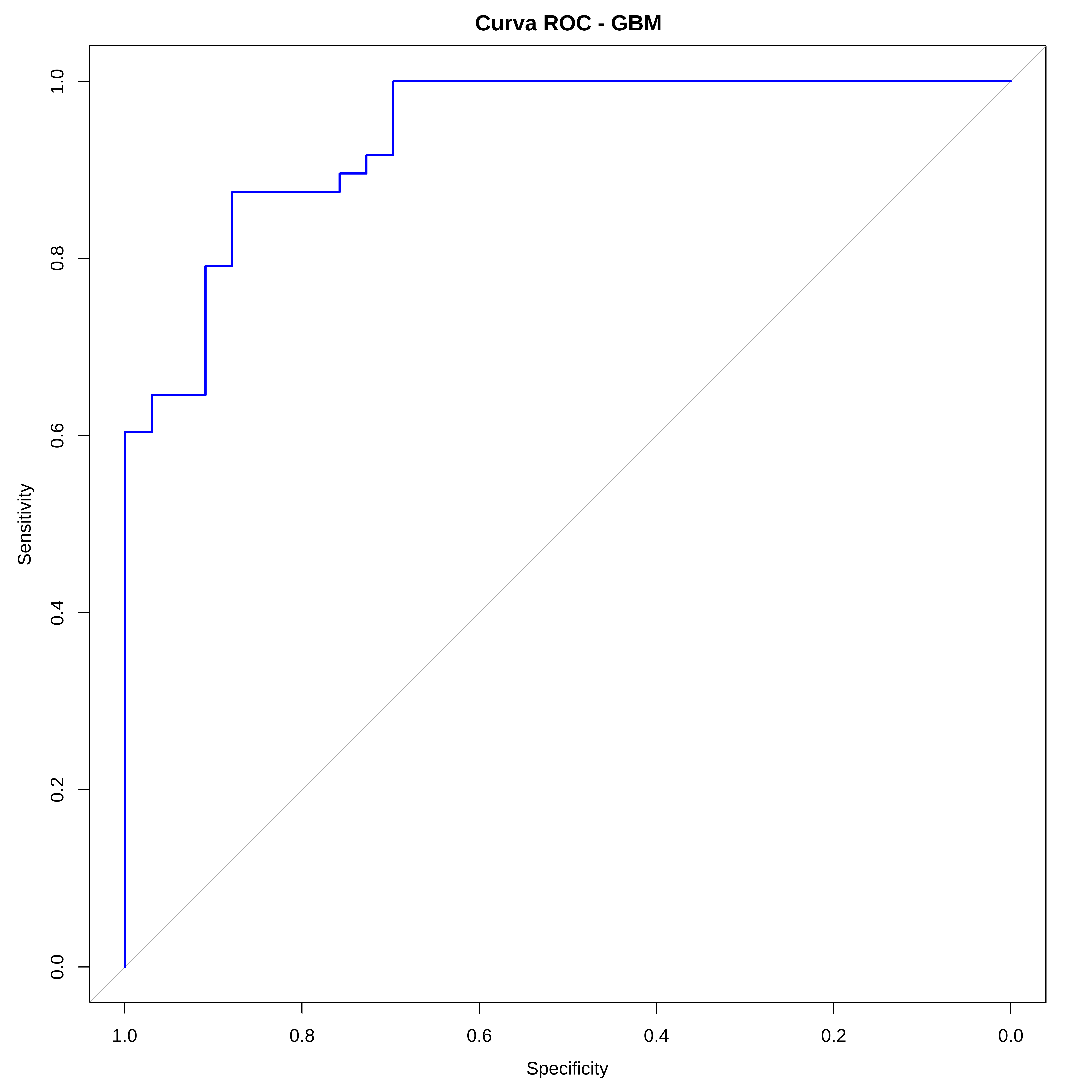
confusionMatrix(pred_class, conj_teste$vendaAlta)Confusion Matrix and Statistics
Reference
Prediction Alta Baixa
Alta 25 6
Baixa 8 42
Accuracy : 0.8272
95% CI : (0.727, 0.9022)
No Information Rate : 0.5926
P-Value [Acc > NIR] : 5.302e-06
Kappa : 0.6386
Mcnemar's Test P-Value : 0.7893
Sensitivity : 0.7576
Specificity : 0.8750
Pos Pred Value : 0.8065
Neg Pred Value : 0.8400
Prevalence : 0.4074
Detection Rate : 0.3086
Detection Prevalence : 0.3827
Balanced Accuracy : 0.8163
'Positive' Class : Alta